Proteazom


Proteazomi su proteinski kompleksi prisutni kod svih eukariota i archaea, i kod nekih bakterija. Kod eukariota, oni se nalaze u jedru i citoplazmi.[1] Glavna funkcija proteazoma je razgradnja nepotrebnih ili oštećenih proteina putem proteolize, hemijske reakcije kojom se razlažu peptidne veze. Enzimi koji izvode ovu reakcije se zovu proteaze. Proteazomi su deo velikog mehanizma kojim ćelije regulišu koncentraciju pojedinih proteina i razgrađuju nepravilno savijene proteine. Proces degradacije proizvodi peptide sa oko sedam do osam aminokiselina, koji se zatim dalje razrađuju u aminokiseline i koriste u sintezi novih proteina.[2] Proteini se obeležavaju za degradaciju malim proteinom zvanim ubikvitin. Reakcija obeležavanja je katalizovana enzimima ubikvitinskim ligazama. Vezivanje jednog ubikvitina na protein je signal drugim ligazama da vežu dodatne molekule ubikvitina. Rezultat je poliubikvitinski lanac za koji se vezuje proteazom, čime se omogućava degradacija ciljnog proteina.[2]
Po strukturi, proteazom je cilindrični kompleks koji sadrži „jezgro” od četiri naslagana prstena koji čine središnju poru. Svaki prsten sačinjen je od sedam pojedinačnih proteina. Unutrašnja dva prstena napravljena su od sedam β podjedinica koje sadrže tri do sedam aktivnih mesta proteaze. Ta mesta nalaze se na unutrašnjoj površini prstenova, tako da ciljni protein mora da uđe u centralnu pora pre nego što se razgradi. Svaka dva spoljna prstena sadrže sedam α podjedinica čija je funkcija održavanje „kapije” kroz koju proteini ulaze u barel. Ove α podjedinice su kontrolisane vezivanjem za „zaklapajuće” strukture ili regulatorne čestice koje prepoznaju poliubikvitinske oznake vezane za proteinske supstrate i pokreću proces razgradnje. Celokupni sistem ubikvitacije i proteazomske razgradnje poznat je pod nazivom ubikvitin-proteazomski sistem.[3]
Put proteazomske razgradnje je važan za mnoge ćelijske procese, uključujući ćelijski ciklus, regulaciju ekspresije gena i response na oksidativni stres. Važnost proteolitičke razgradnje unutar ćelija i uloge ubikvitina u proteolitičkim putevima bila je naglašena dodeljivanjem Nobelove nagrade za hemiju 2004. godine Aronu Čihanoveru, Avramu Heršku i Irvinu Rouzu.[4]
Otkriće[уреди | уреди извор]
Pre otkrića ubikvitinskog proteazomnog sistema, smatralo se da se razgradnja proteina u ćelijama uglavnom oslanja na lizozome, organele vezane za membranu sa kiselim i proteaznim unutrašnjostima, koje mogu da razgrade i potom recikliraju egzogene proteine i stare ili oštećene organele.[2] Međutim, rad Džozefa Etlingera i Alfreda Goldberga iz 1977. godine na ATP-zavisnoj razgradnji proteina u retikulocitima, kojima nedostaju lizozomi, sugerisao je prisustvo drugog mehanizma unutarćelijske degradacije.[5] Za taj proteinski sistem je pokazano 1978. godine da je sačinjen od nekoliko različitih lanca proteina, što je bila novina među proteazama u to vreme.[6] Kasniji rad na modifikaciji histona doveo je do identifikacije neočekivane kovalentne modifikacije proteina histona vezom između lizinskog bočnog lanca histona i C-terminusnog glicinskog ostatka ubikvitina, proteina koji nije imao poznatu funkciju.[7] Zatim je otkriveno da je prethodno identifikovani protein povezan sa proteolitičkom razgradnjom, poznat kao faktor proteolize 1 zavisan od ATP-a (APF-1), isti protein kao i ubikvitin.[8] Proteolitičke aktivnosti ovog sistema izolovane su kao multiproteinski kompleks koji su Šervin Vilk i Marion Orlovski nazvali multikatalitičkim proteinaznim kompleksom.[9] Kasnije je otkriven ATP-zavisni proteolitički kompleks koji je odgovoran za razgradnju proteina zavisnih od ubikvitina i nazvan je 26S proteazom.[10][11]
Veliki deo ranog rada koji je doveo do otkrića ubikvitin proteazomnog sistema odvio se krajem 1970-ih i početkom 1980-ih u Tehnionu u laboratoriji Avrama Herška, gde je Aron Čihanover radio kao postdiplomski student. Herškovo jednogodišnje gostovanje u laboratoriji Irvina Rouza u Foks Čejsovom centru za kancer pružilo je ključne konceptualne spoznaje, mada je Rouz kasnije umanjivao svoju ulogu u otkriću.[12] Njih troje su podelili Nobelovu nagradu za hemiju 2004. godine za njihov rad na otkrivanju ovog sistema.[4]
Elektronsko mikroskopski podaci koji su otkrili prstenastu strukturu naslaganih prstenova proteazoma postali su dostupni sredinom 1980-ih,[13] dok je prva struktura jezgrenog dela proteazoma rešena pomoću rendgenske kristalografije 1994. godine.[14]
Struktura i organizacija[уреди | уреди извор]
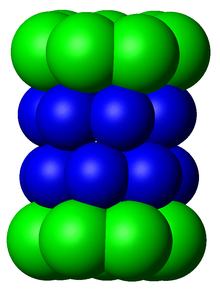
Proteazomske podkomponente često se nazivaju po njihovim Svedbergovim koeficijentima sedimentacije (označenim sa S). Proteazom koji se isključivo koristi kod sisara je citosolni 26S proteasom, koji ima molekulsku masu od oko 2000 kilodaltona (kDa) i sadrži jednu 20S proteinsku podjedinicu i dve 19S regulatorne podjedinice. Jezgro je šuplje i pruža zatvorenu šupljinu u kojoj se proteini razgrađuju; otvori na dva kraja jezgra omogućavaju ulazak ciljnog proteina. Svaki kraj jezgra je udružen sa 19S regulatornom podjedinicom koja sadrži više aktivnih mesta ATPaze i mesta vezanja ubikvitina. Ta struktura prepoznaje poliubikvitinirane proteine i prenosi ih u katalitičko jezgro.[15] Jedan alternativni oblik regulatorne podjedinice, koji se naziva 11S deo, može se povezati s jezgrom na isti način kao i 19S deo. Deo 11S može da igra ulogu u razgradnji stranih peptida, kao što su oni koji nastaju nakon virusne infekcije.[16]
20S deo jezgra[уреди | уреди извор]
Broj i raznolikost podjedinica sadržanih u 20S jezgrenim delovima zavisi od organizma; broj različitih i specijalizovanih podjedinica je veći u višećelijskim nego u jednoćelijskim organizmima, i veći je kod eukariota nego kod prokariota. Svi 20S delovi se sastoje od četiri složene heptamerne prstenaste strukture koje su i same sastavljene od dve različite vrste podjedinica; α podjedinice su strukturne prirode, dok su β podjedinice pretežno katalitičke. α podjedinice su pseudoenzimi koji su homologni β podjedinicama. One su skolopljene sa svojim N-terminusima u blizini onih sa β podjedinica.[17] Spoljašnja dva prstena u snopu se sastoje od sedam α podjedinica koje služe kao domeni za pozicioniranje regulatornih delova, a alfa podjedinični N-terminusi (Pfam PF10584) formiraju kapije koje blokiraju neregulisani pristup supstrata unutrašnjosti šupljine.[18] Unutarnja dva prstena sastoje se od sedam β podjedinica svaki i u njihovim N-terminusima su sadržana aktivna mesta proteaze koja izvode reakcije proteolize.[19] U pročišćenom kompleksu su identifikovane tri različite katalitičke aktivnosti: hidroliza slična himotripsinu, tripsinu i peptidilglutamil-peptidu.[20] Veličina proteazoma je relativno konzervisana i iznosi oko 150 angstrema (Å) sa 115 Å. Unutrašnja komora je široka najviše 53 Å, dok ulaz može biti uzak sa samo 13 Å, što ukazuje na to da se proteini supstrata moraju barem delimično razvući da bi se ušli.[21]
U arhejama kao što je Thermoplasma acidophilum, sve α i sve β podjedinice su identične, dok eukariotski proteazomi poput onih u kvascu sadrže sedam različitih vrsta svake podjedinice. Kod sisara su podjedinice β1, β2 i β5 katalitičke; iako imaju zajednički mehanizam, one imaju tri različite supstratne specifičnosti koje se smatraju sličnim himotripsinu i tripsinu, kao i peptidil-glutamil peptidnoj hidrolizi (PHGH).[22] Alternativni β oblici, koji su označeni sa β1i, β2i i β5i, mogu biti izraženi u hematopoetskim ćelijama kao odgovor na izloženost proinflamatornim signalima, kao što su citokini, naročito interferonu gama. Proteazom sastavljen od ovih alternativnih podjedinica poznat je pod nazivom imunoproteazom, i njegova je specifičnost supstrata izmenjena u odnosu na normalan proteazom.[21] Nedavno je identifikovan alternativni proteazom u ljudskim ćelijama kojima nedostaje α3 podjedinica jezgra.[23] Ovi proteazomi (poznati kao α4-α4 proteazomi) umesto toga formiraju 20S delove jezgra koje sadrže dodatnu α4 podjedinicu umesto nedostajuće α3 podjedinice. Za ove alternativne 'a4-a4' proteazome je ranije bilo poznato da postoje u kvascu.[24] Iako je precizna funkcija ovih proteazomskih izoformi još uvek u velikoj meri nepoznata, ćelije koje izražavaju te proteazome pokazuju pojačanu otpornost na toksičnost indukovanu metalnim jonima, poput kadmijuma.[23][25]
19S regulatorni deo[уреди | уреди извор]
Deo 19S se kod eukariota sastoji se od 19 pojedinačnih proteina i deli se na dva podsklopa, bazu sa 9 podjedinica koja se veže direktno na α prsten jezgrenog dela 20S i poklopac sa 10 podjedinica. Šest od devet proteina baze su ATPazne podjedinice iz porodice AAA, a evolucijski homolog ovih ATPaza postoji kod arheja, koji se naziva PAN (nukleotidaza koja aktivira proteazom).[26] Asocijacija 19S i 20S delova zahteva vezanje ATP-a na podjedinice ATPaze 19S, i neophodna je hidroliza ATP da bi sastavljeni kompleks razgradio sklopljene i ubikvitinisane proteine. Jedino korak rasklapanja supstrata zahteva energiju ATP hidrolize, dok samo vezanje ATP-a može da podržava sve ostale korake potrebne za razgradnju proteina (npr. sklapanje kompleksa, otvaranje kapija, translokacija i proteoliza).[27][28] Zapravo, vezanje ATP-a na ATPaze samo po sebi podržava brzu razgradnju nesklopljenih proteina. Međutim, iako je potrebna hidroliza ATP-a samo za rasklapanje, još nije jasno da li se ta energija možda koristiti u sprezanju nekih od ovih koraka.[28][29]
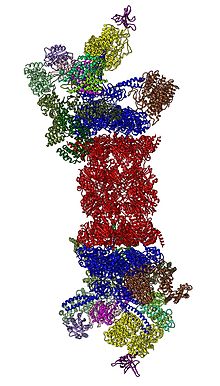
Godine 2012, dva nezavisna napora rasvetlila su molekularnu arhitekturu 26S proteazoma elektronskim mikroskopijom sa jednom česticom.[31][32] Tokom 2016. godine, tri nezavisna napora utvrdila su prvu strukturu u skoro atomskoj rezoluciji ljudskog 26S proteazoma u odsustvu supstrata primenom krio-EM.[33][34][35] U 2018. godini, nakon velikih napora su utvrđeni detaljni mehanizmi deubikvitacije, inicijacije translokacije i procesivnog rasklapanja supstrata, simultanim određivanjem sedam atomskih struktura 26S proteazoma tokom delovanja na supstrat.[15] U srcu 19S-a, neposredno pored 20S-a, nalaze se AAA-ATPaze (AAA proteini) koji se formiraju u heteroheksamerni prsten reda Rpt1/Rpt2/Rpt6/Rpt3/Rpt4/Rpt5. Ovaj prsten je trimer dimera: Rpt1/Rpt2, Rpt6/Rpt3 i Rpt4/Rpt5 se dimerizuju preko njihovih namotanih zavojnica N-terminusa. Ove namotane zavojnice strše iz heksamernog prstena. Najveće regulatorne čestice, ne-ATPaze Rpn1 i Rpn2, vezuju se za vrhove Rpt1/2 i Rpt6/3, respektivno. Ubikvitinski receptor Rpn13 se vezuje za Rpn2 i dovršava bazni potkompleks. Poklopac pokriva polovinu AAA-ATPaze heksamera (Rpt6/Rpt3/Rpt4) i neočekivano direktno je u kontaktu sa 20S preko Rpn6 i u manjem obimu sa Rpn5. Podjedinice Rpn9, Rpn5, Rpn6, Rpn7, Rpn3 i Rpn12, koje su strukturno povezane između sebe i podjedinicama COP9 kompleksa i eIF3 (stoga se nazivaju PCI podjedinice) sastavljaju se u konstrukciju sličnu potkovici koja obuhvata Rpn8/Rpn11 heterodimer. Rpn11, deubikinirajući enzim, smešta se na ulazu AAA-ATPaznog heksamera, idealno je postavljen za uklanjanje ubikvitinskih delova neposredno pre translokacije supstrata u 20S. Drugi ubikvitinski receptor identifikovan do danas, Rpn10, smešten je na obodu poklopca, u blizini podjedinica Rpn8 i Rpn9.
Konformacione promene 19S dela[уреди | уреди извор]
Uočena je 19S regulatorna čestica unutar 26S proteazomskog holoenzima u šest znatno različitih konformacijskih stanja u odsustvu supstrata.[36][37] Prepoznatljivo svojstvo konfiguracije AAA-ATPaze u ovom preovlađujućem stanju niske energije je raspored AAA domena nalik na stepenice.[30][31] U prisustvu ATP-a kad je supstrat odsutan, 19S poprima manje zastupljene konformacije koje se privenstveno razlikuju u poziciji poklopca u odnosu na AAA-ATPazni modul.[33][37] U prisustvu ATP-gS ili supstrata primećeno je znatno više konformacija koje pokazuju dramatične strukturne promene AAA-ATPaznog modula.[15][36][38][39] Neke konformacije sa vezanim supstratom imaju veliku sličnost sa onima bez supstrata, ali nisu potpuno identične, posebno u modulu AAA-ATPaze.[15][36] Pre sklapanja 26S, regulatorna čestica 19S u slobodnom obliku takođe je primećena u sedam konformacijskih stanja.[40] Svi ti konformeri su donekle drugačiji i imaju različite karakteristike. Tako, regulaciona čestica 19S može da poprimi najmanje 20 konformacijskih stanja pod različitim fiziološkim uslovima.
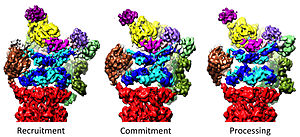
Regulacija 20S pomoću 19S dela[уреди | уреди извор]
Regulatorna čestica 19S odgovorna je za podsticanje 20S na razgradnju proteina. Primarna funkcija regulatornih ATPaza 19S je otvaranje kapije u 20S koja blokira ulazak supstrata u komoru za razgradnju.[41] Nedavno je rasvetljen mehanizam kojim proteazomalna ATPaza otvara ovu kapiju.[18] Za otvaranje 20S vrata i time degradaciju supstrata neophodni su C-termisi proteazomalnih ATPaza, koji sadrže specifičan motiv (tj. HbYX motiv). ATPazni C-terminusi se vežu u otvore u vrhu 20S, i pretvaju ATPazni kompleks u 20S proteolitički kompleks, spajajući na taj način deo za rasklapanje supstrata sa 20S razgradnom mašinom. Vezivanje ovih C-terminusa u ove otvore na 20S samo po sebi podstiče otvaranje kapije u 20S-u na isti način na koji „ključ u bravi” otvara vrata.[18] Precizan način funkcionisanja ovog mehanizma „ključa i brave” je strukturno rasvetljen u kontekstu ljudskog 26S proteazoma do blizu atomske rezolucije, i iz toga proizilazi da je umetanje pet C-terminusa ATPaznih podjedinica Rpt1/2/3/5/6 u otvore na površini 20S neophodno za potpuno otvaranje 20S kapije.[36][15][33]
Drugi regulatorni delovi[уреди | уреди извор]
20S proteazomi se takođe mogu povezati sa drugom vrstom regulatorne čestice, 11S regulatornom česticom, heptamernom strukturom koja ne sadrži ATPaze i može da promoviše razgradnju kratkih peptida, ali ne i kompletnih proteina. Pretpostavlja se da je to zbog toga što kompleks ne može da rasklopi veće supstrate. Ova struktura je takođe poznata kao PA28, REG ili PA26.[17] Mehanizmi pomoću kojih se on veže za jezgro čestica kroz C-terminalne repove svojih podjedinica i indukuje promene konformacija α-prstena za otvaranje kapije 20S sugerirašu sličan mehanizam za česticu 19S.[42] Izražavanje 11S čestice je indukovano interferonom gama i odgovorno je, zajedno sa imunoproteazomskom β podjedinicama, za stvaranje peptida koji se vežu za glavni kompleks histokompatibilnosti.[16]
Još jedan tip ne-ATPaznih regulatornih čestica je Blm10 (kvasac) ili PA200/PSME4 (čovek). On otvara se samo jednu α podjedinicu u kapiji 20S i sam se sklapa u kupolu s vrlo malom porom na vrhu.[17]
Konstrukcija[уреди | уреди извор]
Sastavljanje proteazoma je složen proces zbog broja podjedinica koje se moraju povezati da bi se formirao aktivni kompleks. β podedinice su sintetisane sa N-terminalnim „propeptidima” koji su posttranslaciono modifikovani tokom sastavljanja 20S čestice kako bi se izložilo aktivno proteolitičko mesto. Čestica 20S sastavljena je iz dva poluproteazoma, od kojih se svaki sastoji od sedmočlanog pro-β prstena pričvršćenog na sedmočlani α prsten. Associjacija β-prstenova dva poluproteazoma pokreće autolizu propeptida zavisnu od treonina, kako bi se izložilo aktivno mesto. Ove β interakcije posreduju uglavnom soni mostovi i hidrofobne interakcije između sačuvanih alfa heliksa čiji poremećaj mutacijom onemogućava sposobnost sklapanja proteazoma.[43] Sastavljanje poluprotezoma započinje sastavljanjem α podjedinica u njihov heptamerni prsten, tvoreći šablon za pridruživanje odgovarajućeg pro-β prstena. Sklop α podjedinica nije okarakterisan.[44]
Tek nedavno je proces sastavljanja regulatorne čestice 19S u velikoj meri rasvetljen. Regulatorna čestica 19S formira se kao dve zasebne potkomponente, baza i poklopac. Sastavljanje osnovnog kompleksa je omogućeno pomoću četiri montažna šaperona, Hsm3/S5b, Nas2/p27, Rpn14/PAAF1 i Nas6/gankirin (ova imena su za kvasac/sisare).[45] Ovi montažni šaperoni se vezuju za podjedinice AAA-ATPaze i izgleda da je njihova glavna funkcija obezbeđivanje pravilnog sklapanja heteroheksamernog AAA-ATPaznog prstena. Do danas se još vode rasprave da li se osnovni kompleks sastavlja odvojeno, da li je sklapanje posredovano osnovnom česticom 20S kao šablonom, ili postoje alternativni putevi montaže. Pored četiri montažna šaperona, deubikvitacioni enzim Ubp6/Usp14 takođe pospešuje konstruisanje baze, ali nije esencijalan.[46] Poklopac se sastavlja odvojeno specifičnim redosledom i ne zahteva montažne šaperone.[47]
Proces proteinske degradacije[уреди | уреди извор]
Ubikvitinacija i ciljanje[уреди | уреди извор]
Proteini su ciljani za razgradnju proteazomom putem kovalentne modifikacije lizinskog ostatka koja zahteva koordinirane reakcije tri enzima. U prvom koraku enzim koji aktivira ubikvitin (poznat kao E1) hidrolizuje ATP i adinililuje molekul ubikvitina. Zatim se to prenosi do cisteinskog ostatka aktivnog mesta E1 u kombinaciji sa adenililacijom drugog ubikvitina.[48] Ovaj adinilirani ubikvitin se zatim prenosi na cistein drugog enzima, ubikvitin-konjugirajućeg enzima (E2). U poslednjem koraku, član visoko raznovrsne klase enzima poznatih kao ubikvitinske ligaze (E3) prepoznaje specifični protein koji treba da bude ubikvitiran i katalizuje prenos ubikvitina iz E2 na taj ciljni protein. Ciljni protein mora biti obeležen sa najmanje četiri monomera ubikvitina (u obliku poliubikvitinskog lanca) pre nego što ga poklopac proteazoma može prepoznati.[49] Stoga E3 ovom sistemu daje supstratnu specifičnost.[50] Broj izraženih proteina E1, E2 i E3 zavisi od organizma i ćelijskog tipa. Kod ljudi je prisutno mnogo različitih E3 enzima, što ukazuje na to da postoji ogroman broj meta za ubikvitin proteazomski sistem.
Mehanizam kojim se poliubikvitinirani protein cilja do proteazoma nije u potpunosti razjašnjen. Nekoliko snimaka visoke rezolucije proteazoma vezanog za polubikvitinirani protein sugeriše da receptori ubikvitina mogu biti koordinirani sa deubikvitinazom Rpn11 za početno ciljanje i angažovanje supstrata.[15] Proteini ubikvitinskog receptora imaju N-terminalni domen sličan ubikvitinu (UBL) i jedan ili više domena povezanih sa ubikvitinom (UBA). UBL domene prepoznaju 19S proteazomne kape, i UBA domeni vezuju ubikvitin preko triheliksnog snopa. Ovi receptorski proteini mogu da prate poliubikvitinirane proteine do proteazoma, mada su specifičnosti ove interakcije i njena regulacija nejasni.[51]
Sam protein ubikvitin dugačak je 76 aminokiselina i dobio je ime po svojoj sveprisutnoj prirodi, jer ima visoko očuvanu sekvencu i nalazi se u svim poznatim eukariotskim organizmima.[52] Geni koji kodiraju ubikvitin u eukariotama su raspoređeni u tandemskim ponavljanjima, verovatno zbog velike transkripcione potražnje za ovim genom da se proizvede dovoljno ubikvitina za ćeliju. Predloženo je da je ubikvitin najsporije evoluirajući protein identifikovan do danas.[53] Ubikvitin sadrži sedam ostataka lizina na koje se može ligirati drugi ubikvitin, što rezultira različitim tipovima poliubikvitinskih lanaca.[54] Lanci u kojima je svaki dodatni ubikvitin povezan sa lizinom 48 prethodnog ubikvitina imaju ulogu u proteazomskom ciljanju, dok druge vrste lanaca mogu biti uključene u druge procese.[55][56]

Rasklapanje i translokacija[уреди | уреди извор]
Nakon što je protein ubikvitiniran, prepoznaje ga 19S regulatorna čestica u koraku vezivanja koji zavisi od ATP vezivanja.[15][28] Supstratni protein tada mora da uđe u unutrašnjost 20S čestice da bi došao u kontakt sa proteolitičkim aktivnim mestima. Pošto je središnji kanal čestice 20S sužen i zatvoren N-terminalnim repovima podjedinica α prstena, supstrat mora da poprimi delimično razvijenu konformaciju pre nego što uđu u jezgro.[15] Prelazak nerasklopljenog supstrata u jezgro naziva se translokacija i nužno se dešava nakon deubikvitacije.[15][28] Međutim, još uvek nije jasan redosled u kojem supstrati bivaju deubikvitirani i rasklopljeni.[57] Koji od ovih procesa je korak ograničavanja brzine u celokupnoj reakciji proteolize zavisi od specifičnog supstrata; za neke proteine proces rasklapanja ograničava brzinu, dok je deubikvitacija najsporiji korak za druge proteine.[27] Smatra se da mera u kojoj se supstrati moraju rasklopiti pre translokacije iznosi oko 20 aminokiselinskih ostataka po atomskoj strukturi 26S proteazoma u stanju kompatibilnom s deubikvitacijom,[15] mada je znatna tercijarna struktura, a posebno nelokalne interakcije kao što su disulfidne veze, mogu da budu dovoljne da se inhibira razgradnja.[58] Takođe je predloženo da prisustvo intrinzično neuređenih proteinskih segmenata dovoljne veličine, bilo na kraju proteina ili u unutrašnjosti, može da olakša efikasno započinjanje razgradnje.[59][60]
Kapija formirana od α podjedinica sprečava da peptidi duži od oko četiri ostatka uđu u unutrašnjost 20S čestice. ATP molekuli vezani pre inicijalnog koraka prepoznavanja se hidroliziraju pre translokacije. Dok je energija potrebna za rasklapanje supstrata, ona nije neophodna za translokaciju.[27][28] Formirani 26S proteazom može da razgradi rasklopljene proteine u prisustvu ATP analoga koji se ne hidrolizuje, ali ne može da razgradi sklopljene proteine, što je indikacija da se energija iz hidrolize ATP koristi za rasklapanje supstrata.[27] Do prolaska nerasklopljenog supstrata kroz otvorenu kapiju dolazi putem olakšane difuzije, ako je 19S kapa u stanju vezanom za ATP.[61]
Mehanizam za rasklapanje globularnih proteina je nužno generalan, ali donekle zavisi i od aminokiselinskog niza. Pokazano je da duge sekvence naizmeničnog glicina i alanina inhibiraju rasklapanje supstrata, smanjujući efikasnost proteazomske razgradnje. To rezultira oslobađanjem delimično razgrađenih nusprodukata, verovatno usled rasparivanja hidrolize ATP i koraka rasklapanja.[62] Takva glicin-alaninska ponavljanja se takođe mogu naći u prirodi, na primer u svilenom fibroinu. Specifično, određeni genski proizvodi Epštajn-Barovog virusa koji sadrže ovu sekvencu mogu da zaustave proteazom, pomažući virusu da se razmnoži sprečavanjem prezentacije antigena na glavnom kompleksu histokompatibilnosti.[63]

Proteoliza[уреди | уреди извор]
Mehanizam proteolize pomoću β podjedinica 20S jezgra čestice se odvija putem nukleofilnog napada zavisnog od treonina. Ovaj mehanizam može da zavisi od pridruženog molekula vode za deprotonovanje reaktivnog treoninskog hidroksila. Degradacija se odvija u centralnoj komori formiranoj udruživanjem dva β prstena i obično se ne oslobađaju delimično razgrađeni proizvodi, već se supstrat usitnjava na kratke polipeptide, tipično 7–9 ostataka duge, mada oni mogu biti u opsegu od 4 do 25 ostataka, zavisno od organizma i supstrata. Biohemijski mehanizam koji određuje dužinu proizvoda nije u potpunosti okarakterisan.[64] Iako tri katalitičke β podjedinice imaju zajednički mehanizam, one imaju nešto drugačije specifičnosti supstrata, koje se smatraju sličnim hemotripsinu, tripsinu i peptidil-glutamil-peptid-hidrolizi (PHGH). Ove varijacije specifičnosti rezultat su međusobnih kontakata sa lokalnim ostacima u blizini aktivnih mesta svake podjedinice. Svaka katalitička β podjedinica takođe poseduje konzervirani ostatak lizina koji je neophodan za proteolizu.[22]
Iako proteazom obično proizvodi vrlo kratke fragmente peptida, u nekim slučajevima ovi proizvodi su i biološki aktivni i funkcionalni molekuli. Određeni transkripcioni faktori koji regulišu ekspresiju specifičnih gena, uključujući jednu komponentu sisarskog kompleksa NF-κB, sintetišu se kao neaktivni prekurzori čijom se ubikvitacijom i naknadnom proteazomskom razgradnjom formira aktivna forma. Takva aktivnost zahteva da proteazom preseče unutrašnjost supstratnog proteina, a ne da ga progresivno razgrađuje sa jednog kraja. Smatra se da duge petlje na površinama ovih proteina služe kao proteazomalni supstrati i da ulaze u centralnu šupljinu, dok veći deo proteina ostaje izvan.[65] Slični efekti primećeni su kod kvašćanih proteina; ovaj mehanizam selektivne razgradnje poznat je kao regulirana obrada zavisna od ubikvitina/proteazoma (RUP).[66]
Degradacija nezavisna od ubikvitina[уреди | уреди извор]
Iako većina proteazomalnih supstrata mora da bude ubikvitinirana, pre nego što se razgrade, postoje izuzeci od ovog opšteg pravila, posebno kada proteazom igra normalnu ulogu u posttranslacijskoj obradi proteina. Proteazomalna aktivacija NF-kB preradom p105 u p50 internom proteolizom je jedan od glavnih primera.[65] Neki proteini za koje je hipotetizovano da su nestabilni zbog intrinstično nestrukturiranih regiona,[67] razgrađuju se na način koji je nezavisan od ubikvitina. Najpoznatiji primer proteazomnog supstrata nezavisnog od ubikvitina je enzim ornitin dekarboksilaza.[68] Prijavljeni su i mehanizmi nezavisni od ubikvitina koji ciljaju ključne regulatore ćelijskog ciklusa, kao što je p53, iako je p53 takođe podložan degradaciji koja zavisi od ubikvitina.[69] Konačno, strukturno abnormalni, pogrešno sklopljeni ili visoko oksidovani proteini takođe su podložni degradaciji nezavisnoj od ubikvitina i 19S-nezavisnoj degradaciji u uslovima ćelijskog stresa.[70]
Evolucija[уреди | уреди извор]

Proteazom 20S je sveprisutan i esencijalan kod eukariota. Neki prokarioti, uključujući mnoge arheje i bakterijski red Actinomycetales, takođe imaju homologe 20S proteazoma, dok većina bakterija poseduje gene toplotnog udara hslV i hslU, čiji su genski proizvodi multimerne proteaze raspoređene u dvoslojnom prstenu i ATPaza.[71] Pretpostavlja se da je protein hslV sličan mogućem pretku 20S proteazoma.[72] Generalno, HslV nije esencijalan u bakterijama i nemaju ga sve bakterije, dok neki protisti poseduju 20S i hslV sisteme.[71] Mnoge bakterije takođe poseduju druge homologe proteazoma i pridružene ATPaze, ponajviše ClpP i ClpX. Ova redundantnost objašnjava zašto HslUV sistem nije esencijalan.
Analiza redosleda sekvenci sugeriše da su se katalitičke β podjedinice razišle ranije u evoluciji od pretežno strukturnih α podjedinica. U bakterijama koje izražavaju 20S proteazom, β podjedinice imaju visok nivo sekventne identičnosti sa arhejskim i eukariotskim β podjedinicama, dok je nivo identičnosti α sekvenci mnogo niži. Prisustvo 20S proteazoma u bakterijama može biti rezultat lateralnog prenosa gena, dok se diversifikacija podjedinica među eukariotima pripisuje višestrukim događajima dupliranja gena.[71]
Kontrola ćelijskog ciklusa[уреди | уреди извор]
Progresija ćelijskog ciklusa je regulisana uređenim delovanjem od ciklina zavisnih kinaza (CDK), aktiviranih specifičnim ciklinima koji demarkiraju faze ćelijskog ciklusa. Mitotični ciklini, koji u ćeliji ostaju samo nekoliko minuta, imaju jedan od najkraćih životnih vekova od svih intraćelijskih proteina.[2] Nakon što CDK-ciklinski kompleks izvrši svoju funkciju, pridruženi ciklin se poliubikvitinira i uništava proteazomom, što omogućava usmerenost ćelijskog ciklusa. Konkretno, za izlazak iz mitoze potrebna je od protezoma zavisna disocijacija regulatorne komponente ciklin B od kompleksa faktora promovisanja mitoze.[73] U ćelijama kičmenjaka, „proklizavanje” kroz mitotičku kontrolnu tačku dovodi do prevremenog izlaska iz faze M, uprkos kašnjenja ovog izlaza na kontrolnoj tački vretena.[74]
Ranije kontrolne tačke ćelijskog ciklusa, kao što su provera postrestrikcione tačke između G1 faze i S faze, takođe uključuju proteazomalnu degradaciju ciklina A, čiju ubikvitinaciju promoviše kompleks za promociju anafaze (APC), E3 ubikvitinska ligaza.[75] APC i proteinski kompleks Skp1/Cul1/F-kutija (SCF kompleks) su dva ključna regulatora ciklinske razgradnje i kontrolne tačke; sam SCF se reguliše APC-om preko ubikvitinacije adapterskog proteina, Skp2, što sprečava aktivnost SCF pre G1-S tranzicije.[76]
Pojedinačne komponente 19S čestice imaju svoje regulatorne uloge. Gankirin, nedavno identifikovani onkoprotein, jedan je od 19S potkomponenti koji se takođe čvrsto veže za kinazu zavisnu od ciklina CDK4 i igra ključnu ulogu u prepoznavanju ubikvitinisanog p53, zahvaljujući njegovom afinitetu za ubikvitinsku ligazu MDM2. Gankirin je anti-apoptotičan i pokazalo se da je prekomerno izražen u nekim tipovima ćelija tumora, kao što je hepatocelularni karcinom.[77]
Regulacija biljnog rasta[уреди | уреди извор]
U biljkama, signalizacija pomoću auksina ili fitohormona koji određuju smer i tropizam rasta biljaka, indukuje ciljanje klase represora transkripcionih faktora poznate kao Aux/IAA proteini za proteazomalnu degradaciju. Ovi proteini su ubikvitinisani posredstvom SCFTIR1, ili SCF u kompleksu sa auksinskim receptorom TIR1. Degradacija Aux/IAA proteina derepresuje transkripcione faktore u porodici auksin-responsnih faktora (ARF) i indukuje ARF-usmerenu ekspresiju gena.[78] Ćelijske posledice ARF aktiviranja zavise od tipa biljke i faze razvića, ali su uključene u usmeravanje rasta u korenima i lisnim venama. Smatra se da specifični odgovor na ARF derepresiju posredovan specifičnostima u uparivanju pojedinačnih ARF i Aux/IAA proteina.[79]
Apoptoza[уреди | уреди извор]
Unutrašnji i spoljni signali mogu dovesti do indukcije apoptoze ili programirane ćelijske smrti. Rezultirajuća dekonstrukcija ćelijskih komponenti prvenstveno se vrši posredstvom specijalizovanih proteaza poznatih kao kaspaze, ali proteazom takođe igra važne i raznolike uloge u apoptotičkom procesu. Na uključenost proteazoma u ovaj proces ukazuje povećanje ubikvitacije proteina, i prisustva enzima E1, E2 i E3, što je uočeno i pre apoptoze.[80][81][82] Tokom apoptoze je primećeno da proteazomi lokalizovani u jedru bivaju translocirani na blebove spoljašnje membrane karakteristične za apoptozu.[83]
Inhibicija proteazoma ima različite efekte na indukciju apoptoze u različitim tipovima ćelija. Generalno, proteazom nije neophodan za apoptozu, iako je njegova inhibicija proapoptotička kod većine ćelijskih tipova koji su proučavani. Apoptoza je posredovana prekidom regulisane razgradnje proteina ćelijskog ciklusa rasta.[84] Međutim, neke ćelijske linije - naročito primarne kulture mirujućih i diferenciranih ćelija poput timocita i neurona - bivaju sprečene u podleganju apoptozi izlaganjem proteazomskim inhibitorima. Mehanizam ovog efekta nije jasan, ali se pretpostavlja da je specifičan za ćelije u mirovanju ili da je rezultat diferencijalne aktivnosti proapoptotičke kinaze JNK.[85] Sposobnost proteazomnih inhibitora da indukuju apoptozu u ćelijama koje se brzo dele, iskorišćena je u nekoliko nedavno razvijenih sredstava za hemoterapiju, kao što su bortezomib i salinosporamid A.
Respons na ćelijski stres[уреди | уреди извор]
U responsu na ćelijske stresove - poput infekcije, toplotnog udara ili oksidativnog oštećenja - izražavaju se proteini toplotnog udara koji identifikuju pogrešno sklopljene ili nesklopljene proteine i ciljaju ih za proteazomalnu razgradnju. Hsp27 i Hsp90 su šaperonski proteini koji su povezani sa povećanom aktivnosti ubikvitin-proteazomnog sistema, iako oni nisu direktni učesnici u procesu.[86] S druge strane, Hsp70 veže izložene hidrofobne segmente na površini pogrešno sklopljenih proteina i regrutuje E3 ubikvitinske ligaze, kao što je CHIP, da bi označio proteine za proteazomalnu razgradnju.[87] CHIP protein (karboksilni kraj proteina koji deluje sa Hsp70) sam je regulisan inhibicijom interakcija između E3 enzima CHIP i njegovog E2 vezujućeg partnera.[88]
Slični mehanizmi postoje za promovisanje razgradnje oksidativno oštećenih proteina preko proteazomskog sistema. Proteazomi lokalizovani u jezgru su regulisani PARP-om i aktivno razgrađuju nepodesno oksidovane histone.[89] Oksidovani proteini koji često formiraju velike amorfne agregate u ćeliji mogu se direktno razgraditi pomoću 20S sržnih čestica bez 19S regulacione kape i ne zahtevaju ATP hidrolizu ili obeležavanje ubikvitinom.[70] Međutim, visok nivo oksidativnog oštećenja povećava stupanj umrežavanja proteinskih fragmenata, čineći agregate otpornim na proteolizu. Veći broj i veličina tako visoko oksidovanih agregata povezani su sa starenjem.[90]
Disregulacija ubikvitin proteazomnog sistema može biti izražena u nekoliko neuronskih bolesti. To može dovesti do tumora mozga poput astrocitoma.[91] Kod nekih pozno-nastajućih neurodegenerativnih bolesti koje ispoljavaju nakupljanje pogrešno sklopljenih proteina kao zajedničku karakteristiku, kao što su Parkinsonova bolest i Alchajmerova bolest, veliki nerastvorljivi agregati pogrešno sklopljenih proteina mogu se formirati i potom dovesti do neurotoksičnosti, kroz mehanizme koji još nisu dobro poznati. Smatra se da je smanjena aktivnost proteazoma jedan od uzroka agregacije i formiranja Levijevih tela kod Parkinsona.[92] Ovu hipotezu potkrepljuje zapažanje da su kvaščani modeli Parkinsona podložniji toksičnosti iz α-sinukleina, glavne proteinske komponente Levijevih tela, u uslovima niske aktivnosti proteazoma.[93] Smanjena proteazomalna aktivnost može biti jedan od uzroka kognitivnih poremećaja kao što su poremećaji spektra autizma, i mišićnih i nervnih oboljenja, kao što je inkluziona telesna miopatija.[91]
Uloga u imunskom sistemu[уреди | уреди извор]
Proteazom igra jednostavnu, ali kritičnu ulogu u funkciji adaptivnog imunskog sistema. Peptidni antigeni su prikazani pomoću proteina klase I glavnog kompleksa histokompatibilnosti (MHC) na površini antigen-prezentirajućih ćelija. Ovi peptidi su proizvodi proteazomske razgradnje proteina nastalih od invazivnog patogena. Iako konstitutivno izraženi proteazomi mogu učestvovati u ovom procesu, specijalizovani kompleks sastavljen od proteina čija ekspresija indukuje interferon gama su primarni proizvođači peptida koji su po veličini i sastavu optimalni za vezivanje MHC-a. Ovi proteini čija se ekspresija povećava tokom imunološkog odgovora uključuju 11S regulatornu česticu, čija je glavna poznata biološka uloga regulisanje proizvodnje MHC liganda, i specijalizovanih β podjedinica zvanih β1i, β2i, i β5i sa izmenjenom supstratnom specifičnošću. Kompleks formiran sa specijalizovanim β podjedinicama poznat je kao imunoproteazom.[16] Druga varijanta β5i, podjedinica β5t, izražena je u timusu, što dovodi do specifičnog za timus „tireoproteazoma” čija je funkcija još uvek nejasna.[94]
Jačina vezivanja liganda MHC klase I zavisi od sastava ligandovog C-kraja, jer se peptidi vezuju vodoničnim vezivanjem i bliskim kontaktima sa regionom zvanim „B džep” na MHC površini. Mnogi aleli MHC klase I preferiraju hidrofobne C-terminalne ostatke, a imunoproteazomski kompleks je verovatniji da stvara hidrofobne C-terminise.[95]
Zbog svoje uloge u stvaranju aktiviranog oblika NF-κB, antiapoptotičkog i proupalnog regulatora ekspresije citokina, proteazomalna aktivnost je povezana sa upalnim i autoimunskim bolestima. Povećani nivoi proteazomske aktivnosti koreliraju sa aktivnostima bolesti i povezani su sa autoimunskim bolestima uključujući sistemski eritematozni lupus i reumatoidni artritis.[16]
Proteazom je takođe uključen u unutarćelijsku antitelom-posredovanu proteolizu viriona vezanih za antitelo. Na ovom neutralizacionom putu, TRIM21 (protein iz porodice tripartitnih motiva) se veže sa imunoglobulinom G da bi usmerio virion u proteazom, gde biva razgrađen.[96]
Proteazomski inhibitori[уреди | уреди извор]


Proteazomski inhibitori ispoljavaju efektivno anti-tumorsko dejstvo u ćelijskoj kulturi, indukujući apoptozu ometanjem regulisane razgradnje proteina ćelijskog ciklusa rasta.[84] Ovakav pristup selektivne indukcije apoptoze u ćelijama tumora pokazao se efikasnim u životinjskim modelima i u kliničkim ispitivanjima.
Laktacistin, prirodni proizvod koji sintetiše bakterija Streptomyces, bio je prvi nepeptidni proteazomni inhibitor koji je otkriven[97] i koji se široko koristi kao istraživačko sredstvo u biohemiji i ćelijskoj biologiji. Laktacistin je licenciralo preduzeće Miogeniks/Proskript, koje je otkupila firma Milenijum Farmaceutikals, sada deo Takede. Laktacistin kovalentno modifikuje amino-terminalni treonin katalitičke β podjedinice proteazoma, naročito β5 podjedinicu koja je odgovorna za proteazomsku aktivnost sličnu himotripsinu. Ovo otkriće pomoglo je da se proteazom uspostavi kao mehanistički nova klasa proteaze: amino-terminalna treoninska proteaza.
Bortezomib (borirani MG132), molekul koji je razvilo preduzeće Milenijum Farmaceutikals i koji se prodaje kao Velkad, prvi je proteazomski inhibitor koji je našao kliničku primenu kao hemoterapijsko sredstvo.[98] Bortezomib se koristi u tretmanu multiplog mijeloma.[99] Za multipli mijelom je uočeno da dovodi do povećanih nivoa peptida formiranih dejstvom proteazoma u kvrnom serumu. Koncentracije tih peptida se smanjuju na normalne nivoe kao odgovor na uspešnu hemoterapiju.[100] Studije na životinjama pokazale su da bortezomib takođe može imati klinički značajne efekte kod raka pankreasa.[101][102] Predklinička i rana klinička ispitivanja započela su radi ispitivanja efikasnosti bortezomiba u lečenju drugih karcinoma povezanih sa B-ćelijama,[103] posebno nekih tipova nehodžkinovog limfoma.[104] Klinički rezultati isto tako opravdavaju upotrebu proteazomskih inhibitora u kombinaciji sa hemoterapijom, za akutnu limfoblastičnu leukemiju B-ćelija.[105] Proteazomni inhibitori mogu usmrtiti neke tipove kultivisanih ćelija leukemije koje su rezistentne na glukokortikoide.[106]
Molekul ritonavir, u prodaji kao Norvir, razvijen je kao proteazni inhibitor i korišten je za tretiranje HIV infekcije. Međutim, pokazalo se da inhibira proteazome kao i slobodne proteaze; specifičnije, ritonavir inhibira proteazomnu aktivnost koja je nalik na himotripsin, dok je aktivnost slična tripsinu donekle pojačana.[107] Studije na životinjskim modelima ukazuju na to da ritonavir može da ima inhibitorni efekat na rast ćelija glioma.[108]
Proteazomni inhibitori su takođe pokazali obećavajuće rezultate u lečenju autoimunih bolesti na životinjskim modelima. Na primer, studije na miševima sa presađenom ljudskom kožom pokazale su smanjenje veličine lezija usled psorijaze nakon lečenja proteazomnim inhibitorom.[109] Inhibitori takođe pokazuju pozitivne efekte u glodarskim modelima astme.[110]
Obeležavanje i inhibicija proteazoma takođe je od interesa za laboratorijske postavke i za in vitro i in vivo istraživanje proteazomske aktivnosti u ćelijama. Najčešće korišćeni laboratorijski inhibitori su laktacistin i peptidni aldehid MG132 koji je inicijalno razvila laboratorija Goldberg. Fluorescentni inhibitori su takođe razvijeni da specifično obeležavaju aktivna mesta sklopljenog proteazoma.[111]
Klinički značaj[уреди | уреди извор]
Proteazom i njegove podjedinice su od kliničkog značaja iz najmanje dva razloga: (1) kompromitovani kompleksni sklop ili disfunkcionalni proteazom mogu biti povezani sa osnovom patofiziologije specifičnih bolesti, i (2) oni se mogu koristiti kao mete za lekove u terapeutskim intervencijama. U novije vreme uloženo je više napora da se razmotri proteazom za razvoj novih dijagnostičkih markera i strategija. Poboljšano i sveobuhvatno razumevanje patofiziologije proteazoma trebalo bi da dovede do kliničkih primena u budućnosti.
Proteazomi formiraju središnju komponentu ubikuitin-proteazomnog sistema (UPS)[112] i odgovarajuće ćelijske kontrole kvaliteta proteina (PKC). Ubikvitacija proteina i naknadna proteoliza i razgradnja proteazomom su važni mehanizmi u regulaciji ćelijskog ciklusa, ćelijskom rastu i diferencijaciji, transkripciji gena, transdukciji signala i apoptozi.[113] Nakon toga, kompromitovani kompleks i funkcija proteazoma dovode do smanjenja proteolitičkih aktivnosti i nakupljanja oštećenih ili pogrešno sklopljenih proteina. Takvo nagomilavanje proteina može doprineti patogenezi i fenotipskim karakteristikama neurodegenerativnih bolesti,[114][115] kardiovaskularnim bolestima,[116][117][118] upalnim odgovorima i autoimunskim bolestima,[119] i sistemskim odgovorima oštećenja DNK koja dovode do malignih oboljenja.[120]
Nekoliko eksperimentalnih i kliničkih studija je pokazalo da aberacije i deregulacije UPS doprinose patogenezi nekoliko neurodegenerativnih i miodegenerativnih poremećaja, uključujući Alchajmerovu bolest,[121] Parkinsonovu bolest[122] i Pikovu bolest,[123] amiotofičnu lateralnu sklerozu (ALS),[123] Hantingtonovu bolest,[122] Krojcfeldt-Jakobovu bolest,[124] i motorno neuronske bolesti, poliglutaminske (PolyQ) bolesti, mišićne distrofije[125] i nekoliko retkih oblika neurodegenerativnih bolesti povezanih sa demencijom.[126] Kao deo ubikvitin-proteazomnog sistema (UPS), proteazom održava srčanu proteinsku homeostazu i na taj način igra značajnu ulogu u srčanom ishemijskom oštećenju,[127] ventrikularnoj hipertrofiji[128] i zatajenju srca.[129] Uz to, gomilaju se dokazi da UPS igra ključnu ulogu u malignoj transformaciji. UPS proteoliza igra veliku ulogu u odgovorima ćelija raka na stimulativne signale koji su kritični za razvoj raka. Shodno tome, ekspresija gena degradacijom transkripcionih faktora, kao što su p53, c-Jun, c-Fos, NF-κB, c-Myc, HIF-1α, MATα2, STAT3, sterolom regulisani proteini vezivanja elemenata i androgeni receptori su svi kontrolisani putem UPS-a i na taj način uključeni u razvoj raznih maligniteta.[130] Štaviše, UPS reguliše razgradnju tumorsko supresorskih gena kao što je adenomatozna polipoza koli (APC) kod kolorektalnog karcinoma, retinoblastoma (Rb) i fon Hipel-Lindau supresora tumora (VHL), kao i niz proto-onkogena (Raf, Myc, Myb, Rel, Src, Mos, Abl). UPS je takođe uključen u regulaciju upalnih reakcija. Ova aktivnost se obično pripisuje ulozi proteazoma u aktivaciji NF-κB, čime se dalje reguliše ekspresija proupalnih citokina poput TNF-α, IL-β, IL-8, adhezivnih molekula (ICAM-1, VCAM-1, P-selektin), prostaglandini i azotni oksid (NO).[119] Pored toga, UPS takođe igra ulogu u upalnim reakcijama kao regulatorima proliferacije leukocita, uglavnom proteolizom ciklina i degradacijom CDK inhibitora.[131] Konačno, pacijenti sa autoimunskom bolešću sa SLE, Šegrenovim sindromom i reumatoidnim artritisom (RA) uglavnom pokazuju cirkulišuće proteazome koji se mogu primeniti kao klinički biomarkeri.[132]
Vidi još[уреди | уреди извор]
Reference[уреди | уреди извор]
- ^ Peters, Jan-Michael; Franke, Werner W.; Kleinschmidt, Jiirgen A. (1994). „Distinct 19 S and 20 S subcomplexes of the 26 S proteasome and their distribution in the nucleus and the cytoplasm”. The Journal of Biological Chemistry. 269 (10): 7709—18. PMID 8125997. Архивирано из оригинала 24. 02. 2020. г. Приступљено 26. 10. 2012.
- ^ а б в г Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J (2004). „3”. Molecular cell biology (5th изд.). New York: W.H. Freeman and CO. стр. 66—72. ISBN 0-7167-4366-3.
- ^ Nassif, Nicholas D.; Cambray, Samantha E.; Kraut, Daniel A. (2014). „Slipping up: Partial substrate degradation by ATP-dependent proteases”. IUBMB Life. 66 (5): 309—317. PMID 24823973. doi:10.1002/iub.1271.
- ^ а б Nobel Prize Committee (2004). „Nobel Prize Awardees in Chemistry, 2004”. Приступљено 2006-12-11.
- ^ Etlinger JD, Goldberg AL (1977). „A soluble ATP-dependent proteolytic system responsible for the degradation of abnormal proteins in reticulocytes”. Proceedings of the National Academy of Sciences of the United States of America. 74 (1): 54—8. PMC 393195
 . PMID 264694. doi:10.1073/pnas.74.1.54.
. PMID 264694. doi:10.1073/pnas.74.1.54.
- ^ Ciehanover A, Hod Y, Hershko A (1978). „A heat-stable polypeptide component of an ATP-dependent proteolytic system from reticulocytes”. Biochemical and Biophysical Research Communications. 81 (4): 1100—5. PMID 666810. doi:10.1016/0006-291X(78)91249-4.
- ^ Goldknopf IL, Busch H (1977). „Isopeptide linkage between nonhistone and histone 2A polypeptides of chromosomal conjugate-protein A24”. Proceedings of the National Academy of Sciences of the United States of America. 74 (3): 864—8. PMC 430507
 . PMID 265581. doi:10.1073/pnas.74.3.864.
. PMID 265581. doi:10.1073/pnas.74.3.864.
- ^ Ciechanover A (2005). „Early work on the ubiquitin proteasome system, an interview with Aaron Ciechanover. Interview by CDD”. Cell Death and Differentiation. 12 (9): 1167—77. PMID 16094393. doi:10.1038/sj.cdd.4401691.
- ^ Wilk S, Orlowski M (1980). „Cation-sensitive neutral endopeptidase: isolation and specificity of the bovine pituitary enzyme”. Journal of Neurochemistry. 35 (5): 1172—82. PMID 6778972. doi:10.1111/j.1471-4159.1980.tb07873.x.
- ^ Arrigo AP, Tanaka, K, Goldberg F, Welch WJ (1988). „Identity of 19S prosome particle with the large multifunctional protease complex of mammalian cells.”. Nature. 331 (6152): 192—94. PMID 3277060. doi:10.1038/331192a0.Tanaka K, Waxman L, Goldberg AL (1983). „ATP serves two distinct roles in protein degradation in reticulocytes, one requiring and one independent of ubiquitin”. The Journal of Cell Biology. 96 (6): 1580—5. PMC 2112434
 . PMID 6304111. doi:10.1083/jcb.96.6.1580.
. PMID 6304111. doi:10.1083/jcb.96.6.1580.
- ^ Hough R, Pratt G, Rechsteiner M (1987). „Purification of two high molecular weight proteases from rabbit reticulocyte lysate”. The Journal of Biological Chemistry. 262 (17): 8303—13. PMID 3298229.[мртва веза]
- ^ Hershko A (2005). „Early work on the ubiquitin proteasome system, an interview with Avram Hershko. Interview by CDD”. Cell Death and Differentiation. 12 (9): 1158—61. PMID 16094391. doi:10.1038/sj.cdd.4401709.
- ^ Kopp F, Steiner R, Dahlmann B, Kuehn L, Reinauer H (1986). „Size and shape of the multicatalytic proteinase from rat skeletal muscle”. Biochimica et Biophysica Acta. 872 (3): 253—60. PMID 3524688. doi:10.1016/0167-4838(86)90278-5.
- ^ Löwe J, Stock D, Jap B, Zwickl P, Baumeister W, Huber R (1995). „Crystal structure of the 20S proteasome from the archaeon T. acidophilum at 3.4 A resolution”. Science. 268 (5210): 533—9. PMID 7725097. doi:10.1126/science.7725097.
- ^ а б в г д ђ е ж з и Dong Y, Zhang S, Wu Z, Li X, Wang WL, Zhu Y, Stoilova-McPhie S, Lu Y, Finley D, Mao Y (2018). „Cryo-EM structures and dynamics of substrate-engaged human 26S proteasome”. Nature. 565 (7737): 49—55. PMC 6370054
 . PMID 30479383. doi:10.1038/s41586-018-0736-4.
. PMID 30479383. doi:10.1038/s41586-018-0736-4.
- ^ а б в г Wang J, Maldonado MA (2006). „The ubiquitin-proteasome system and its role in inflammatory and autoimmune diseases”. Cellular & Molecular Immunology. 3 (4): 255—61. PMID 16978533.
- ^ а б в Stadtmueller, BM; Hill, CP (2011-01-07). „Proteasome activators.”. Molecular Cell. 41 (1): 8—19. PMC 3040445
 . PMID 21211719. doi:10.1016/j.molcel.2010.12.020.
. PMID 21211719. doi:10.1016/j.molcel.2010.12.020.
- ^ а б в Smith DM, Chang SC, Park S, Finley D, Cheng Y, Goldberg AL (2007). „Docking of the proteasomal ATPases' carboxyl termini in the 20S proteasome's alpha ring opens the gate for substrate entry”. Molecular Cell. 27 (5): 731—44. PMC 2083707
 . PMID 17803938. doi:10.1016/j.molcel.2007.06.033.
. PMID 17803938. doi:10.1016/j.molcel.2007.06.033.
- ^ „MEROPS Family T1”. EMBL-EBI. Приступљено 2019-02-16.
- ^ Wilk S, Orlowski M (1983). „Evidence that pituitary cation-sensitive neutral endopeptidase is a multicatalytic protease complex”. Journal of Neurochemistry. 40 (3): 842—9. PMID 6338156. S2CID 23508675. doi:10.1111/j.1471-4159.1983.tb08056.x.
- ^ а б Nandi D, Tahiliani P, Kumar A, Chandu D (2006). „The ubiquitin-proteasome system” (PDF). Journal of Biosciences. 31 (1): 137—55. PMID 16595883. S2CID 21603835. doi:10.1007/BF02705243.
- ^ а б Heinemeyer W, Fischer M, Krimmer T, Stachon U, Wolf DH (1997). „The active sites of the eukaryotic 20 S proteasome and their involvement in subunit precursor processing”. The Journal of Biological Chemistry. 272 (40): 25200—9. PMID 9312134. doi:10.1074/jbc.272.40.25200
 .
.
- ^ а б Padmanabhan A, Vuong SA, Hochstrasser M (2016). „Assembly of an Evolutionarily Conserved Alternative Proteasome Isoform in Human Cells”. Cell Reports. 14 (12): 2962—74. PMC 4828729
 . PMID 26997268. doi:10.1016/j.celrep.2016.02.068.
. PMID 26997268. doi:10.1016/j.celrep.2016.02.068.
- ^ Velichutina I, Connerly PL, Arendt CS, Li X, Hochstrasser M (2004). „Plasticity in eucaryotic 20S proteasome ring assembly revealed by a subunit deletion in yeast”. The EMBO Journal. 23 (3): 500—10. PMC 1271798
 . PMID 14739934. doi:10.1038/sj.emboj.7600059.
. PMID 14739934. doi:10.1038/sj.emboj.7600059.
- ^ Kusmierczyk AR, Kunjappu MJ, Funakoshi M, Hochstrasser M (2008). „A multimeric assembly factor controls the formation of alternative 20S proteasomes”. Nature Structural & Molecular Biology. 15 (3): 237—44. PMID 18278055. S2CID 21181637. doi:10.1038/nsmb.1389.
- ^ Zwickl P, Ng D, Woo KM, Klenk HP, Goldberg AL (1999). „An archaebacterial ATPase, homologous to ATPases in the eukaryotic 26 S proteasome, activates protein breakdown by 20 S proteasomes”. The Journal of Biological Chemistry. 274 (37): 26008—14. PMID 10473546. doi:10.1074/jbc.274.37.26008
 .
.
- ^ а б в г Smith DM, Kafri G, Cheng Y, Ng D, Walz T, Goldberg AL (2005). „ATP binding to PAN or the 26S ATPases causes association with the 20S proteasome, gate opening, and translocation of unfolded proteins”. Molecular Cell. 20 (5): 687—98. PMID 16337593. doi:10.1016/j.molcel.2005.10.019.
- ^ а б в г д Liu CW, Li X, Thompson D, Wooding K, Chang TL, Tang Z, Yu H, Thomas PJ, DeMartino GN (2006). „ATP binding and ATP hydrolysis play distinct roles in the function of 26S proteasome”. Molecular Cell. 24 (1): 39—50. PMC 3951175
 . PMID 17018291. doi:10.1016/j.molcel.2006.08.025.
. PMID 17018291. doi:10.1016/j.molcel.2006.08.025.
- ^ Lam YA, Lawson TG, Velayutham M, Zweier JL, Pickart CM (2002). „A proteasomal ATPase subunit recognizes the polyubiquitin degradation signal”. Nature. 416 (6882): 763—7. Bibcode:2002Natur.416..763L. PMID 11961560. S2CID 4421764. doi:10.1038/416763a.
- ^ а б Beck F, Unverdorben P, Bohn S, Schweitzer A, Pfeifer G, Sakata E, Nickell S, Plitzko JM, Villa E, Baumeister W, Förster F (2012). „Near-atomic resolution structural model of the yeast 26S proteasome”. Proceedings of the National Academy of Sciences of the United States of America. 109 (37): 14870—5. Bibcode:2012PNAS..10914870B. PMC 3443124
 . PMID 22927375. doi:10.1073/pnas.1213333109
. PMID 22927375. doi:10.1073/pnas.1213333109  .
.
- ^ а б Lander GC, Estrin E, Matyskiela ME, Bashore C, Nogales E, Martin A (2012). „Complete subunit architecture of the proteasome regulatory particle”. Nature. 482 (7384): 186—91. Bibcode:2012Natur.482..186L. PMC 3285539
 . PMID 22237024. doi:10.1038/nature10774.
. PMID 22237024. doi:10.1038/nature10774.
- ^ Lasker K, Förster F, Bohn S, Walzthoeni T, Villa E, Unverdorben P, Beck F, Aebersold R, Sali A, Baumeister W (2012). „Molecular architecture of the 26S proteasome holocomplex determined by an integrative approach”. Proceedings of the National Academy of Sciences of the United States of America. 109 (5): 1380—7. PMC 3277140
 . PMID 22307589. doi:10.1073/pnas.1120559109
. PMID 22307589. doi:10.1073/pnas.1120559109  .
.
- ^ а б в Chen S, Wu J, Lu Y, Ma YB, Lee BH, Yu Z, Ouyang Q, Finley DJ, Kirschner MW, Mao Y (2016). „Structural basis for dynamic regulation of the human 26S proteasome”. Proceedings of the National Academy of Sciences of the United States of America. 113 (46): 12991—12996. Bibcode:2016PNAS..11312991C. PMC 5135334
 . PMID 27791164. doi:10.1073/pnas.1614614113
. PMID 27791164. doi:10.1073/pnas.1614614113  .
.
- ^ Huang X, Luan B, Wu J, Shi Y (2016). „An atomic structure of the human 26S proteasome”. Nature Structural & Molecular Biology. 23 (9): 778—785. PMID 27428775. S2CID 21909333. doi:10.1038/nsmb.3273.
- ^ Schweitzer A, Aufderheide A, Rudack T, Beck F, Pfeifer G, Plitzko JM, Sakata E, Schulten K, Förster F, Baumeister W (2016). „Structure of the human 26S proteasome at a resolution of 3.9 Å”. Proceedings of the National Academy of Sciences of the United States of America. 113 (28): 7816—7821. Bibcode:2016PNAS..11312991C. PMC 5135334
 . PMID 27791164. doi:10.1073/pnas.1614614113
. PMID 27791164. doi:10.1073/pnas.1614614113  .
.
- ^ а б в г Zhu Y, Wang WL, Yu D, Ouyang Q, Lu Y, Mao Y (2018). „Structural mechanism for nucleotide-driven remodeling of the AAA-ATPase unfoldase in the activated human 26S proteasome”. Nature Communications. 9 (1): 1360. Bibcode:2018NatCo...9.1360Z. PMC 5893597
 . PMID 29636472. doi:10.1038/s41467-018-03785-w.
. PMID 29636472. doi:10.1038/s41467-018-03785-w.
- ^ а б в Unverdorben P, Beck F, Śledź P, Schweitzer A, Pfeifer G, Plitzko JM, Baumeister W, Förster F (2014). „Deep classification of a large cryo-EM dataset defines the conformational landscape of the 26S proteasome”. Proceedings of the National Academy of Sciences of the United States of America. 111 (15): 5544—9. Bibcode:2014PNAS..111.5544U. PMC 3992697
 . PMID 24706844. doi:10.1073/pnas.1403409111
. PMID 24706844. doi:10.1073/pnas.1403409111  .
.
- ^ Śledź P, Unverdorben P, Beck F, Pfeifer G, Schweitzer A, Förster F, Baumeister W (2013). „Structure of the 26S proteasome with ATP-γS bound provides insights into the mechanism of nucleotide-dependent substrate translocation”. Proceedings of the National Academy of Sciences of the United States of America. 110 (18): 7264—7269. Bibcode:2013PNAS..110.7264S. PMC 3645540
 . PMID 23589842. doi:10.1073/pnas.1305782110
. PMID 23589842. doi:10.1073/pnas.1305782110  .
.
- ^ Matyskiela ME, Lander GC, Martin A (2013). „Conformational switching of the 26S proteasome enables substrate degradation”. Nature Structural & Molecular Biology. 20 (7): 781—788. PMC 3712289
 . PMID 23770819. doi:10.1038/nsmb.2616.
. PMID 23770819. doi:10.1038/nsmb.2616.
- ^ Lu Y, Wu J, Dong Y, Chen S, Sun S, Ma YB, Ouyang Q, Finley D, Kirschner MW, Mao Y (2017). „Conformational Landscape of the p28-Bound Human Proteasome Regulatory Particle”. Molecular Cell. 67 (2): 322—333.e6. PMC 5580496
 . PMID 28689658. doi:10.1016/j.molcel.2017.06.007.
. PMID 28689658. doi:10.1016/j.molcel.2017.06.007.
- ^ Köhler A, Cascio P, Leggett DS, Woo KM, Goldberg AL, Finley D (2001). „The axial channel of the proteasome core particle is gated by the Rpt2 ATPase and controls both substrate entry and product release”. Molecular Cell. 7 (6): 1143—52. PMID 11430818. doi:10.1016/S1097-2765(01)00274-X.
- ^ Förster A, Masters EI, Whitby FG, Robinson H, Hill CP (2005). „The 1.9 A structure of a proteasome-11S activator complex and implications for proteasome-PAN/PA700 interactions”. Molecular Cell. 18 (5): 589—99. PMID 15916965. doi:10.1016/j.molcel.2005.04.016.
- ^ Witt S, Kwon YD, Sharon M, Felderer K, Beuttler M, Robinson CV, Baumeister W, Jap BK (2006). „Proteasome assembly triggers a switch required for active-site maturation”. Structure. 14 (7): 1179—88. PMID 16843899. doi:10.1016/j.str.2006.05.019.
- ^ Krüger E, Kloetzel PM, Enenkel C (2001). „20S proteasome biogenesis”. Biochimie. 83 (3–4): 289—93. PMID 11295488. doi:10.1016/S0300-9084(01)01241-X.
- ^ Murata S, Yashiroda H, Tanaka K (2009). „Molecular mechanisms of proteasome assembly”. Nature Reviews Molecular Cell Biology. 10 (2): 104—115. PMID 19165213. doi:10.1038/nrm2630.
- ^ Sakata E, Stengel F, Fukunaga K, Zhou M, Saeki Y, Förster F, Baumeister W, Tanaka K, Robinson CV (2011). „The catalytic activity of Ubp6 enhances maturation of the proteasomal regulatory particle”. Molecular Cell. 42 (5): 637—649. PMID 21658604. doi:10.1016/j.molcel.2011.04.021.
- ^ Fukunaga K, Kudo T, Toh-e A, Tanaka K, Saeki Y (2010). „Dissection of the assembly pathway of the proteasome lid in Saccharomyces cerevisiae”. Biochemical and Biophysical Research Communications. 396 (4): 1048—1053. PMID 20471955. doi:10.1016/j.bbrc.2010.05.061.
- ^ Haas AL, Warms JV, Hershko A, Rose IA (1982). „Ubiquitin-activating enzyme. Mechanism and role in protein-ubiquitin conjugation”. The Journal of Biological Chemistry. 257 (5): 2543—8. PMID 6277905.
- ^ Thrower JS, Hoffman L, Rechsteiner M, Pickart CM (2000). „Recognition of the polyubiquitin proteolytic signal”. The EMBO Journal. 19 (1): 94—102. PMC 1171781
 . PMID 10619848. doi:10.1093/emboj/19.1.94.
. PMID 10619848. doi:10.1093/emboj/19.1.94.
- ^ Risseeuw EP, Daskalchuk TE, Banks TW, Liu E, Cotelesage J, Hellmann H, Estelle M, Somers DE, Crosby WL (2003). „Protein interaction analysis of SCF ubiquitin E3 ligase subunits from Arabidopsis”. The Plant Journal. 34 (6): 753—67. PMID 12795696. doi:10.1046/j.1365-313X.2003.01768.x.
- ^ Elsasser S, Finley D (2005). „Delivery of ubiquitinated substrates to protein-unfolding machines”. Nature Cell Biology. 7 (8): 742—9. PMID 16056265. doi:10.1038/ncb0805-742.
- ^ Sadanandom A, Bailey M, Ewan R, Lee J, Nelis S (2012). „The ubiquitin-proteasome system: central modifier of plant signalling”. The New Phytologist. 196 (1): 13—28. PMID 22897362. doi:10.1111/j.1469-8137.2012.04266.x.
- ^ Sharp PM, Li WH (1987). „Ubiquitin genes as a paradigm of concerted evolution of tandem repeats”. Journal of Molecular Evolution. 25 (1): 58—64. PMID 3041010. doi:10.1007/BF02100041.
- ^ Pickart CM, Fushman D (2004). „Polyubiquitin chains: polymeric protein signals”. Current Opinion in Chemical Biology. 8 (6): 610—16. PMID 15556404. doi:10.1016/j.cbpa.2004.09.009.
- ^ Xu P, Duong DM, Seyfried NT, Cheng D, Xie Y, Robert J, Rush J, Hochstrasser M, Finley D, Peng J (2009). „Quantitative proteomics reveals the function of unconventional ubiquitin chains in proteasomal degradation”. Cell. 137 (1): 133—45. PMC 2668214
 . PMID 19345192. doi:10.1016/j.cell.2009.01.041.
. PMID 19345192. doi:10.1016/j.cell.2009.01.041.
- ^ Pickart CM (2000). „Ubiquitin in chains”. Trends in Biochemical Sciences. 25 (11): 544—8. PMID 11084366. doi:10.1016/S0968-0004(00)01681-9.
- ^ Zhu Q, Wani G, Wang QE, El-mahdy M, Snapka RM, Wani AA (2005). „Deubiquitination by proteasome is coordinated with substrate translocation for proteolysis in vivo”. Experimental Cell Research. 307 (2): 436—51. PMID 15950624. doi:10.1016/j.yexcr.2005.03.031.
- ^ Wenzel T, Baumeister W (1995). „Conformational constraints in protein degradation by the 20S proteasome”. Nature Structural Biology. 2 (3): 199—204. PMID 7773788. doi:10.1038/nsb0395-199.
- ^ Inobe T, Fishbain S, Prakash S, Matouschek A (2011). „Defining the geometry of the two-component proteasome degron”. Nature Chemical Biology. 7 (3): 161—7. PMC 3129032
 . PMID 21278740. doi:10.1038/nchembio.521.
. PMID 21278740. doi:10.1038/nchembio.521.
- ^ van der Lee R, Lang B, Kruse K, Gsponer J, Sánchez de Groot N, Huynen MA, Matouschek A, Fuxreiter M, Babu MM (2014). „Intrinsically disordered segments affect protein half-life in the cell and during evolution”. Cell Reports. 8 (6): 1832—44. PMC 4358326
 . PMID 25220455. doi:10.1016/j.celrep.2014.07.055.
. PMID 25220455. doi:10.1016/j.celrep.2014.07.055.
- ^ Smith DM, Benaroudj N, Goldberg A (2006). „Proteasomes and their associated ATPases: a destructive combination”. Journal of Structural Biology. 156 (1): 72—83. PMID 16919475. doi:10.1016/j.jsb.2006.04.012.
- ^ Hoyt MA, Zich J, Takeuchi J, Zhang M, Govaerts C, Coffino P (2006). „Glycine-alanine repeats impair proper substrate unfolding by the proteasome”. The EMBO Journal. 25 (8): 1720—9. PMC 1440830
 . PMID 16601692. doi:10.1038/sj.emboj.7601058.
. PMID 16601692. doi:10.1038/sj.emboj.7601058.
- ^ Zhang M, Coffino P (2004). „Repeat sequence of Epstein–Barr virus-encoded nuclear antigen 1 protein interrupts proteasome substrate processing”. The Journal of Biological Chemistry. 279 (10): 8635—41. PMID 14688254. doi:10.1074/jbc.M310449200.
- ^ Voges D, Zwickl P, Baumeister W (1999). „The 26S proteasome: a molecular machine designed for controlled proteolysis”. Annual Review of Biochemistry. 68 (1): 1015—68. PMID 10872471. doi:10.1146/annurev.biochem.68.1.1015.
- ^ а б Rape M, Jentsch S (2002). „Taking a bite: proteasomal protein processing”. Nature Cell Biology. 4 (5): E113—6. PMID 11988749. doi:10.1038/ncb0502-e113.
- ^ Rape M, Jentsch S (2004). „Productive RUPture: activation of transcription factors by proteasomal processing”. Biochimica et Biophysica Acta. 1695 (1–3): 209—13. PMID 15571816. doi:10.1016/j.bbamcr.2004.09.022.
- ^ Asher G, Reuven N, Shaul Y (2006). „20S proteasomes and protein degradation "by default"”. BioEssays. 28 (8): 844—9. PMID 16927316. doi:10.1002/bies.20447.
- ^ Zhang M, Pickart CM, Coffino P (2003). „Determinants of proteasome recognition of ornithine decarboxylase, a ubiquitin-independent substrate”. The EMBO Journal. 22 (7): 1488—96. PMC 152902
 . PMID 12660156. doi:10.1093/emboj/cdg158.
. PMID 12660156. doi:10.1093/emboj/cdg158.
- ^ Asher G, Shaul Y (2005). „p53 proteasomal degradation: poly-ubiquitination is not the whole story”. Cell Cycle. 4 (8): 1015—8. PMID 16082197. doi:10.4161/cc.4.8.1900.
- ^ а б Shringarpure R, Grune T, Mehlhase J, Davies KJ (2003). „Ubiquitin conjugation is not required for the degradation of oxidized proteins by proteasome”. The Journal of Biological Chemistry. 278 (1): 311—8. PMID 12401807. doi:10.1074/jbc.M206279200.
- ^ а б в Gille C, Goede A, Schlöetelburg C, Preissner R, Kloetzel PM, Göbel UB, Frömmel C (2003). „A comprehensive view on proteasomal sequences: implications for the evolution of the proteasome”. Journal of Molecular Biology. 326 (5): 1437—48. PMID 12595256. doi:10.1016/S0022-2836(02)01470-5.
- ^ Bochtler M, Ditzel L, Groll M, Hartmann C, Huber R (1999). „The proteasome”. Annual Review of Biophysics and Biomolecular Structure. 28 (1): 295—317. PMID 10410804. doi:10.1146/annurev.biophys.28.1.295.
- ^ Chesnel F, Bazile F, Pascal A, Kubiak JZ (2006). „Cyclin B dissociation from CDK1 precedes its degradation upon MPF inactivation in mitotic extracts of Xenopus laevis embryos”. Cell Cycle. 5 (15): 1687—98. PMID 16921258. doi:10.4161/cc.5.15.3123.
- ^ Brito DA, Rieder CL (2006). „Mitotic checkpoint slippage in humans occurs via cyclin B destruction in the presence of an active checkpoint”. Current Biology. 16 (12): 1194—200. PMC 2749311
 . PMID 16782009. doi:10.1016/j.cub.2006.04.043.
. PMID 16782009. doi:10.1016/j.cub.2006.04.043.
- ^ Havens CG, Ho A, Yoshioka N, Dowdy SF (2006). „Regulation of late G1/S phase transition and APC Cdh1 by reactive oxygen species”. Molecular and Cellular Biology. 26 (12): 4701—11. PMC 1489138
 . PMID 16738333. doi:10.1128/MCB.00303-06.
. PMID 16738333. doi:10.1128/MCB.00303-06.
- ^ Bashir T, Dorrello NV, Amador V, Guardavaccaro D, Pagano M (2004). „Control of the SCF(Skp2-Cks1) ubiquitin ligase by the APC/C(Cdh1) ubiquitin ligase”. Nature. 428 (6979): 190—3. PMID 15014502. doi:10.1038/nature02330.
- ^ Higashitsuji H, Liu Y, Mayer RJ, Fujita J (2005). „The oncoprotein gankyrin negatively regulates both p53 and RB by enhancing proteasomal degradation”. Cell Cycle. 4 (10): 1335—7. PMID 16177571. doi:10.4161/cc.4.10.2107.
- ^ Dharmasiri S, Estelle M (2002). „The role of regulated protein degradation in auxin response”. Plant Molecular Biology. 49 (3–4): 401—9. PMID 12036263. doi:10.1023/A:1015203013208.
- ^ Weijers D, Benkova E, Jäger KE, Schlereth A, Hamann T, Kientz M, Wilmoth JC, Reed JW, Jürgens G (2005). „Developmental specificity of auxin response by pairs of ARF and Aux/IAA transcriptional regulators”. The EMBO Journal. 24 (10): 1874—85. PMC 1142592
 . PMID 15889151. doi:10.1038/sj.emboj.7600659.
. PMID 15889151. doi:10.1038/sj.emboj.7600659.
- ^ Haas AL, Baboshina O, Williams B, Schwartz LM (1995). „Coordinated induction of the ubiquitin conjugation pathway accompanies the developmentally programmed death of insect skeletal muscle”. The Journal of Biological Chemistry. 270 (16): 9407—12. PMID 7721865. doi:10.1074/jbc.270.16.9407.
- ^ Schwartz LM, Myer A, Kosz L, Engelstein M, Maier C (1990). „Activation of polyubiquitin gene expression during developmentally programmed cell death”. Neuron. 5 (4): 411—9. PMID 2169771. doi:10.1016/0896-6273(90)90080-Y.
- ^ Löw P, Bussell K, Dawson SP, Billett MA, Mayer RJ, Reynolds SE (1997). „Expression of a 26S proteasome ATPase subunit, MS73, in muscles that undergo developmentally programmed cell death, and its control by ecdysteroid hormones in the insect Manduca sexta”. FEBS Letters. 400 (3): 345—9. PMID 9009228. doi:10.1016/S0014-5793(96)01413-5.
- ^ Pitzer F, Dantes A, Fuchs T, Baumeister W, Amsterdam A (1996). „Removal of proteasomes from the nucleus and their accumulation in apoptotic blebs during programmed cell death”. FEBS Letters. 394 (1): 47—50. PMID 8925925. doi:10.1016/0014-5793(96)00920-9.
- ^ а б Adams J, Palombella VJ, Sausville EA, Johnson J, Destree A, Lazarus DD, Maas J, Pien CS, Prakash S, Elliott PJ (1999). „Proteasome inhibitors: a novel class of potent and effective antitumor agents”. Cancer Research. 59 (11): 2615—22. PMID 10363983.
- ^ Orlowski RZ (1999). „The role of the ubiquitin-proteasome pathway in apoptosis”. Cell Death and Differentiation. 6 (4): 303—13. PMID 10381632. doi:10.1038/sj.cdd.4400505.
- ^ Garrido C, Brunet M, Didelot C, Zermati Y, Schmitt E, Kroemer G (2006). „Heat shock proteins 27 and 70: anti-apoptotic proteins with tumorigenic properties”. Cell Cycle. 5 (22): 2592—601. PMID 17106261. doi:10.4161/cc.5.22.3448.
- ^ Park SH, Bolender N, Eisele F, Kostova Z, Takeuchi J, Coffino P, Wolf DH (2007). „The cytoplasmic Hsp70 chaperone machinery subjects misfolded and endoplasmic reticulum import-incompetent proteins to degradation via the ubiquitin-proteasome system”. Molecular Biology of the Cell. 18 (1): 153—65. PMC 1751312
 . PMID 17065559. doi:10.1091/mbc.E06-04-0338.
. PMID 17065559. doi:10.1091/mbc.E06-04-0338.
- ^ Dai Q, Qian SB, Li HH, McDonough H, Borchers C, Huang D, Takayama S, Younger JM, Ren HY, Cyr DM, Patterson C (2005). „Regulation of the cytoplasmic quality control protein degradation pathway by BAG2”. The Journal of Biological Chemistry. 280 (46): 38673—81. PMID 16169850. doi:10.1074/jbc.M507986200.
- ^ Bader N, Grune T (2006). „Protein oxidation and proteolysis”. Biological Chemistry. 387 (10–11): 1351—5. PMID 17081106. doi:10.1515/BC.2006.169.
- ^ Davies KJ (2003). „Degradation of oxidized proteins by the 20S proteasome”. Biochimie. 83 (3–4): 301—10. PMID 11295490. doi:10.1016/S0300-9084(01)01250-0.
- ^ а б Lehman NL (2009). „The ubiquitin proteasome system in neuropathology”. Acta Neuropathologica. 118 (3): 329—47. PMC 2716447
 . PMID 19597829. doi:10.1007/s00401-009-0560-x.
. PMID 19597829. doi:10.1007/s00401-009-0560-x.
- ^ McNaught KS, Jackson T, JnoBaptiste R, Kapustin A, Olanow CW (2006). „Proteasomal dysfunction in sporadic Parkinson's disease”. Neurology. 66 (10 Suppl 4): S37—49. PMID 16717251. doi:10.1212/01.wnl.0000221745.58886.2e.
- ^ Sharma N, Brandis KA, Herrera SK, Johnson BE, Vaidya T, Shrestha R, Debburman SK (2006). „alpha-Synuclein budding yeast model: toxicity enhanced by impaired proteasome and oxidative stress”. Journal of Molecular Neuroscience. 28 (2): 161—78. PMID 16679556. doi:10.1385/JMN:28:2:161.
- ^ Murata S, Sasaki K, Kishimoto T, Niwa S, Hayashi H, Takahama Y, Tanaka K (2007). „Regulation of CD8+ T cell development by thymus-specific proteasomes”. Science. 316 (5829): 1349—53. PMID 17540904. doi:10.1126/science.1141915.
- ^ Cascio P, Hilton C, Kisselev AF, Rock KL, Goldberg AL (2001). „26S proteasomes and immunoproteasomes produce mainly N-extended versions of an antigenic peptide”. The EMBO Journal. 20 (10): 2357—66. PMC 125470
 . PMID 11350924. doi:10.1093/emboj/20.10.2357.
. PMID 11350924. doi:10.1093/emboj/20.10.2357.
- ^ Mallery DL, McEwan WA, Bidgood SR, Towers GJ, Johnson CM, James LC (2010). „Antibodies mediate intracellular immunity through tripartite motif-containing 21 (TRIM21)”. Proceedings of the National Academy of Sciences of the United States of America. 107 (46): 19985—19990. PMC 2993423
 . PMID 21045130. doi:10.1073/pnas.1014074107. Архивирано из оригинала 06. 09. 2019. г. Приступљено 07. 09. 2019.
. PMID 21045130. doi:10.1073/pnas.1014074107. Архивирано из оригинала 06. 09. 2019. г. Приступљено 07. 09. 2019.
- ^ Fenteany G, Standaert RF, Lane WS, Choi S, Corey EJ, Schreiber SL (1995). „Inhibition of proteasome activities and subunit-specific amino-terminal threonine modification by lactacystin”. Science. 268 (5211): 726—31. PMID 7732382. doi:10.1126/science.7732382.
- ^ United States Food and Drug Administration press release Архивирано 2007-02-19 на сајту Wayback Machine 13 May 2003. Access date 29 December 2006. See also FDA Velcade information page.
- ^ Fisher RI, Bernstein SH, Kahl BS, Djulbegovic B, Robertson MJ, de Vos S, Epner E, Krishnan A, Leonard JP, Lonial S, Stadtmauer EA, O'Connor OA, Shi H, Boral AL, Goy A (2006). „Multicenter phase II study of bortezomib in patients with relapsed or refractory mantle cell lymphoma”. Journal of Clinical Oncology. 24 (30): 4867—74. PMID 17001068. doi:10.1200/JCO.2006.07.9665.
- ^ Jakob C, Egerer K, Liebisch P, Türkmen S, Zavrski I, Kuckelkorn U, Heider U, Kaiser M, Fleissner C, Sterz J, Kleeberg L, Feist E, Burmester GR, Kloetzel PM, Sezer O (2007). „Circulating proteasome levels are an independent prognostic factor for survival in multiple myeloma”. Blood. 109 (5): 2100—5. PMID 17095627. doi:10.1182/blood-2006-04-016360.
- ^ Shah SA, Potter MW, McDade TP, Ricciardi R, Perugini RA, Elliott PJ, Adams J, Callery MP (2001). „26S proteasome inhibition induces apoptosis and limits growth of human pancreatic cancer”. Journal of Cellular Biochemistry. 82 (1): 110—22. PMID 11400168. doi:10.1002/jcb.1150.
- ^ Nawrocki ST, Sweeney-Gotsch B, Takamori R, McConkey DJ (2004). „The proteasome inhibitor bortezomib enhances the activity of docetaxel in orthotopic human pancreatic tumor xenografts”. Molecular Cancer Therapeutics. 3 (1): 59—70. PMID 14749476.
- ^ Schenkein D (2002). „Proteasome inhibitors in the treatment of B-cell malignancies”. Clinical Lymphoma. 3 (1): 49—55. PMID 12141956. doi:10.3816/CLM.2002.n.011.
- ^ O'Connor OA, Wright J, Moskowitz C, Muzzy J, MacGregor-Cortelli B, Stubblefield M, Straus D, Portlock C, Hamlin P, Choi E, Dumetrescu O, Esseltine D, Trehu E, Adams J, Schenkein D, Zelenetz AD (2005). „Phase II clinical experience with the novel proteasome inhibitor bortezomib in patients with indolent non-Hodgkin's lymphoma and mantle cell lymphoma”. Journal of Clinical Oncology. 23 (4): 676—84. PMID 15613699. doi:10.1200/JCO.2005.02.050.
- ^ Messinger YH, Gaynon PS, Sposto R, van der Giessen J, Eckroth E, Malvar J, Bostrom BC (2012). „Bortezomib with chemotherapy is highly active in advanced B-precursor acute lymphoblastic leukemia: Therapeutic Advances in Childhood Leukemia & Lymphoma (TACL) Study”. Blood. 120 (2): 285—90. PMID 22653976. doi:10.1182/blood-2012-04-418640.
- ^ Lambrou GI, Papadimitriou L, Chrousos GP, Vlahopoulos SA (2012). „Glucocorticoid and proteasome inhibitor impact on the leukemic lymphoblast: multiple, diverse signals converging on a few key downstream regulators”. Molecular and Cellular Endocrinology. 351 (2): 142—51. PMID 22273806. doi:10.1016/j.mce.2012.01.003.
- ^ Schmidtke G, Holzhütter HG, Bogyo M, Kairies N, Groll M, de Giuli R, Emch S, Groettrup M (1999). „How an inhibitor of the HIV-I protease modulates proteasome activity”. The Journal of Biological Chemistry. 274 (50): 35734—40. PMID 10585454. doi:10.1074/jbc.274.50.35734.
- ^ Laurent N, de Boüard S, Guillamo JS, Christov C, Zini R, Jouault H, Andre P, Lotteau V, Peschanski M (2004). „Effects of the proteasome inhibitor ritonavir on glioma growth in vitro and in vivo”. Molecular Cancer Therapeutics. 3 (2): 129—36. PMID 14985453.
- ^ Zollner TM, Podda M, Pien C, Elliott PJ, Kaufmann R, Boehncke WH (2002). „Proteasome inhibition reduces superantigen-mediated T cell activation and the severity of psoriasis in a SCID-hu model”. The Journal of Clinical Investigation. 109 (5): 671—9. PMC 150886
 . PMID 11877475. doi:10.1172/JCI12736.
. PMID 11877475. doi:10.1172/JCI12736.
- ^ Elliott PJ, Pien CS, McCormack TA, Chapman ID, Adams J (1999). „Proteasome inhibition: A novel mechanism to combat asthma”. The Journal of Allergy and Clinical Immunology. 104 (2 Pt 1): 294—300. PMID 10452747. doi:10.1016/S0091-6749(99)70369-6.
- ^ Verdoes M, Florea BI, Menendez-Benito V, Maynard CJ, Witte MD, van der Linden WA, van den Nieuwendijk AM, Hofmann T, Berkers CR, van Leeuwen FW, Groothuis TA, Leeuwenburgh MA, Ovaa H, Neefjes JJ, Filippov DV, van der Marel GA, Dantuma NP, Overkleeft HS (2006). „A fluorescent broad-spectrum proteasome inhibitor for labeling proteasomes in vitro and in vivo”. Chemistry & Biology. 13 (11): 1217—26. PMID 17114003. doi:10.1016/j.chembiol.2006.09.013.
- ^ Kleiger G, Mayor T (2014). „Perilous journey: a tour of the ubiquitin-proteasome system”. Trends in Cell Biology. 24 (6): 352—9. PMC 4037451
 . PMID 24457024. doi:10.1016/j.tcb.2013.12.003.
. PMID 24457024. doi:10.1016/j.tcb.2013.12.003.
- ^ Goldberg AL, Stein R, Adams J (1995). „New insights into proteasome function: from archaebacteria to drug development”. Chemistry & Biology. 2 (8): 503—8. PMID 9383453. doi:10.1016/1074-5521(95)90182-5.
- ^ Sulistio YA, Heese K (2015). „The Ubiquitin-Proteasome System and Molecular Chaperone Deregulation in Alzheimer's Disease”. Molecular Neurobiology. 53 (2): 905—31. PMID 25561438. doi:10.1007/s12035-014-9063-4.
- ^ Ortega Z, Lucas JJ (2014). „Ubiquitin-proteasome system involvement in Huntington's disease”. Frontiers in Molecular Neuroscience. 7: 77. PMC 4179678
 . PMID 25324717. doi:10.3389/fnmol.2014.00077.
. PMID 25324717. doi:10.3389/fnmol.2014.00077.
- ^ Sandri M, Robbins J (2014). „Proteotoxicity: an underappreciated pathology in cardiac disease”. Journal of Molecular and Cellular Cardiology. 71: 3—10. PMC 4011959
 . PMID 24380730. doi:10.1016/j.yjmcc.2013.12.015.
. PMID 24380730. doi:10.1016/j.yjmcc.2013.12.015.
- ^ Drews O, Taegtmeyer H (2014). „Targeting the ubiquitin-proteasome system in heart disease: the basis for new therapeutic strategies”. Antioxidants & Redox Signaling. 21 (17): 2322—43. PMC 4241867
 . PMID 25133688. doi:10.1089/ars.2013.5823.
. PMID 25133688. doi:10.1089/ars.2013.5823.
- ^ Wang ZV, Hill JA (2015). „Protein quality control and metabolism: bidirectional control in the heart”. Cell Metabolism. 21 (2): 215—26. PMC 4317573
 . PMID 25651176. doi:10.1016/j.cmet.2015.01.016.
. PMID 25651176. doi:10.1016/j.cmet.2015.01.016.
- ^ а б Karin M, Delhase M (2000). „The I kappa B kinase (IKK) and NF-kappa B: key elements of proinflammatory signalling”. Seminars in Immunology. 12 (1): 85—98. PMID 10723801. doi:10.1006/smim.2000.0210.
- ^ Ermolaeva MA, Dakhovnik A, Schumacher B (2015). „Quality control mechanisms in cellular and systemic DNA damage responses”. Ageing Research Reviews. 23 (Pt A): 3—11. PMC 4886828
 . PMID 25560147. doi:10.1016/j.arr.2014.12.009.
. PMID 25560147. doi:10.1016/j.arr.2014.12.009.
- ^ Checler F, da Costa CA, Ancolio K, Chevallier N, Lopez-Perez E, Marambaud P (2000). „Role of the proteasome in Alzheimer's disease”. Biochimica et Biophysica Acta. 1502 (1): 133—8. PMID 10899438. doi:10.1016/s0925-4439(00)00039-9.
- ^ а б Chung KK, Dawson VL, Dawson TM (2001). „The role of the ubiquitin-proteasomal pathway in Parkinson's disease and other neurodegenerative disorders”. Trends in Neurosciences. 24 (11 Suppl): S7—14. PMID 11881748. doi:10.1016/s0166-2236(00)01998-6.
- ^ а б Ikeda K, Akiyama H, Arai T, Ueno H, Tsuchiya K, Kosaka K (2002). „Morphometrical reappraisal of motor neuron system of Pick's disease and amyotrophic lateral sclerosis with dementia”. Acta Neuropathologica. 104 (1): 21—8. PMID 12070660. doi:10.1007/s00401-001-0513-5.
- ^ Manaka H, Kato T, Kurita K, Katagiri T, Shikama Y, Kujirai K, Kawanami T, Suzuki Y, Nihei K, Sasaki H (1992). „Marked increase in cerebrospinal fluid ubiquitin in Creutzfeldt–Jakob disease”. Neuroscience Letters. 139 (1): 47—9. PMID 1328965. doi:10.1016/0304-3940(92)90854-z.
- ^ Mathews KD, Moore SA (2003). „Limb-girdle muscular dystrophy”. Current Neurology and Neuroscience Reports. 3 (1): 78—85. PMID 12507416. doi:10.1007/s11910-003-0042-9.
- ^ Mayer RJ (2003). „From neurodegeneration to neurohomeostasis: the role of ubiquitin”. Drug News & Perspectives. 16 (2): 103—8. PMID 12792671. doi:10.1358/dnp.2003.16.2.829327.
- ^ Calise J, Powell SR (2013). „The ubiquitin proteasome system and myocardial ischemia”. American Journal of Physiology. Heart and Circulatory Physiology. 304 (3): H337—49. PMC 3774499
 . PMID 23220331. doi:10.1152/ajpheart.00604.2012.
. PMID 23220331. doi:10.1152/ajpheart.00604.2012.
- ^ Predmore JM, Wang P, Davis F, Bartolone S, Westfall MV, Dyke DB, Pagani F, Powell SR, Day SM (2010). „Ubiquitin proteasome dysfunction in human hypertrophic and dilated cardiomyopathies”. Circulation. 121 (8): 997—1004. PMC 2857348
 . PMID 20159828. doi:10.1161/CIRCULATIONAHA.109.904557.
. PMID 20159828. doi:10.1161/CIRCULATIONAHA.109.904557.
- ^ Powell SR (2006). „The ubiquitin-proteasome system in cardiac physiology and pathology”. American Journal of Physiology. Heart and Circulatory Physiology. 291 (1): H1—H19. PMID 16501026. doi:10.1152/ajpheart.00062.2006.
- ^ Adams J (2003). „Potential for proteasome inhibition in the treatment of cancer”. Drug Discovery Today. 8 (7): 307—15. PMID 12654543. doi:10.1016/s1359-6446(03)02647-3.
- ^ Ben-Neriah Y (2002). „Regulatory functions of ubiquitination in the immune system”. Nature Immunology. 3 (1): 20—6. PMID 11753406. doi:10.1038/ni0102-20.
- ^ Egerer K, Kuckelkorn U, Rudolph PE, Rückert JC, Dörner T, Burmester GR, Kloetzel PM, Feist E (2002). „Circulating proteasomes are markers of cell damage and immunologic activity in autoimmune diseases”. The Journal of Rheumatology. 29 (10): 2045—52. PMID 12375310.
Literatura[уреди | уреди извор]
- Glickman, Michael H.; Adir, Noam (2004). „The Proteasome and the Delicate Balance between Destruction and Rescue”. PLoS Biology. 2 (1): e13. PMC 314468
 . PMID 14737189. doi:10.1371/journal.pbio.0020013.
. PMID 14737189. doi:10.1371/journal.pbio.0020013. - The Yeast 26S Proteasome with list of subunits and pictures
- Ciechanover, A (2005). „Early work on the ubiquitin proteasome system, an interview with Aaron Ciechanover”. Cell Death and Differentiation. 12 (9): 1167—77. PMID 16094393. doi:10.1038/sj.cdd.4401691.
- Hershko, A (2005). „Early work on the ubiquitin proteasome system, an interview with Avram Hershko”. Cell Death and Differentiation. 12 (9): 1158—61. PMID 16094391. doi:10.1038/sj.cdd.4401709.
- Adams, J (2005). „Early work on the ubiquitin proteasome system, an interview with Irwin Rose”. Cell Death and Differentiation. 12 (9): 1162—6. PMID 16094392. doi:10.1038/sj.cdd.4401700.
- Cvek, B; Dvorak, Z (2007). „Targeting of nuclear factor-kappaB and proteasome by dithiocarbamate complexes with metals”. Current pharmaceutical design. 13 (30): 3155—67. PMID 17979756. doi:10.2174/138161207782110390. Архивирано из оригинала 29. 07. 2012. г. Приступљено 25. 08. 2018.
- Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J (2004). „3”. Molecular cell biology (5th изд.). New York: W.H. Freeman and CO. стр. 66—72. ISBN 0-7167-4366-3.
Spoljašnje veze[уреди | уреди извор]
