Дезоксирибонуклеинска киселина — разлика између измена
| Ред 187: | Ред 187: | ||
[[File:Benzopyrene DNA adduct 1JDG.png|thumb|right|[[Ковалентна веза|Ковалентни]] додатак [[Citohrom P450, familija 1, ćlan A1|метаболички активиране]] форме [[Benzo(a)piren|бензо[''а'']пирена]], главног [[мутаген]]а [[Duvanski dim|дуванског дима]], у ДНК ({{PDB2|1JDG}})<ref>{{cite journal |title = Solution structure of a trans-opened (10S)-dA adduct of (+)-(7S,8R,9S,10R)-7,8-dihydroxy-9,10-epoxy-7,8,9,10-tetrahydrobenzo[a]pyrene in a fully complementary DNA duplex: evidence for a major syn conformation |author = Padmanava Pradhan, Sampath Tirumala, Xiaohong Liu, Jane M. Sayer, Donald M. Jerina, and Herman J. C. Yeh |journal = Biochemistry |year = 2001 |volume = 40 |issue = 20 |pages = 5870–5881 |doi = 10.1021/bi002896q |url = http://pubs.acs.org/doi/abs/10.1021/bi002896q}}</ref><ref>{{cite journal |title = NMR Solution Structure of a Nonanucleotide Duplex with a dG Mismatch Opposite a 10R Adduct Derived from Trans Addition of a Deoxyadenosine N6-Amino Group to (-)-(7S,8R,9R,10S)-7,8-Dihydroxy-9,10-epoxy-7,8,9,10-tetrahydrobenzo[a]pyrene |author = Eric J. Schurter, Herman J. C. Yeh, Jane M. Sayer, Mahesh K. Lakshman, Haruhiko Yagi, Donald M. Jerina, David G. Gorenstein |journal = Biochemistry |year = 1995 |volume = 34 |issue = 4 |pages = 1364–1375 |doi = 10.1021/bi00004a031 |url = http://pubs.acs.org/doi/abs/10.1021/bi00004a031}}</ref><ref>{{cite journal |title = NMR Evidence for Syn-Anti Interconversion of a Trans Opened (10R)-dA Adduct of Benzo[a]pyrene (7S,8R)-Diol (9R,10S)-Epoxide in a DNA Duplex |author = D. E. Volk, J. S. Rice, B. A. Luxon, H. J. C. Yeh, C. Liang, G. Xie, J. M. Sayer, D. M. Jerina, and D. G. Gorenstein |journal = Biochemistry |year = 2000 |volume = 39 |issue = 46, |pages = 14040–14053 |doi = 10.1021/bi001669l |url = http://pubs.acs.org/doi/abs/10.1021/bi001669l}}</ref>]] |
[[File:Benzopyrene DNA adduct 1JDG.png|thumb|right|[[Ковалентна веза|Ковалентни]] додатак [[Citohrom P450, familija 1, ćlan A1|метаболички активиране]] форме [[Benzo(a)piren|бензо[''а'']пирена]], главног [[мутаген]]а [[Duvanski dim|дуванског дима]], у ДНК ({{PDB2|1JDG}})<ref>{{cite journal |title = Solution structure of a trans-opened (10S)-dA adduct of (+)-(7S,8R,9S,10R)-7,8-dihydroxy-9,10-epoxy-7,8,9,10-tetrahydrobenzo[a]pyrene in a fully complementary DNA duplex: evidence for a major syn conformation |author = Padmanava Pradhan, Sampath Tirumala, Xiaohong Liu, Jane M. Sayer, Donald M. Jerina, and Herman J. C. Yeh |journal = Biochemistry |year = 2001 |volume = 40 |issue = 20 |pages = 5870–5881 |doi = 10.1021/bi002896q |url = http://pubs.acs.org/doi/abs/10.1021/bi002896q}}</ref><ref>{{cite journal |title = NMR Solution Structure of a Nonanucleotide Duplex with a dG Mismatch Opposite a 10R Adduct Derived from Trans Addition of a Deoxyadenosine N6-Amino Group to (-)-(7S,8R,9R,10S)-7,8-Dihydroxy-9,10-epoxy-7,8,9,10-tetrahydrobenzo[a]pyrene |author = Eric J. Schurter, Herman J. C. Yeh, Jane M. Sayer, Mahesh K. Lakshman, Haruhiko Yagi, Donald M. Jerina, David G. Gorenstein |journal = Biochemistry |year = 1995 |volume = 34 |issue = 4 |pages = 1364–1375 |doi = 10.1021/bi00004a031 |url = http://pubs.acs.org/doi/abs/10.1021/bi00004a031}}</ref><ref>{{cite journal |title = NMR Evidence for Syn-Anti Interconversion of a Trans Opened (10R)-dA Adduct of Benzo[a]pyrene (7S,8R)-Diol (9R,10S)-Epoxide in a DNA Duplex |author = D. E. Volk, J. S. Rice, B. A. Luxon, H. J. C. Yeh, C. Liang, G. Xie, J. M. Sayer, D. M. Jerina, and D. G. Gorenstein |journal = Biochemistry |year = 2000 |volume = 39 |issue = 46, |pages = 14040–14053 |doi = 10.1021/bi001669l |url = http://pubs.acs.org/doi/abs/10.1021/bi001669l}}</ref>]] |
||
ДНК може да буде оштећена многим врстама [[мутаген]]а, који мењању ДНК секвенцу. Мутагени обухватају [[Oksidacioni angens|оксидационе агенсе]], [[Alkilacija|алкилирајуће агенсе]], као и [[Електромагнетско зрачење|електромагнетну радијацију]] високе енергије, попут [[Ultraljubičasto zračenje|ултраљубичастог]] светла и [[Rendgenski zraci|X-зрака]]. Тип произведеног ДНК оштећења зависи од типа мутагена. На пример, УВ светло може да оштети ДНК формирањем [[Pirimidinski dimer|тиминских димера]], који су међусобно повезани између пиримидинских база.<ref>{{cite journal |author = Douki T, Reynaud-Angelin A, Cadet J, Sage E |title = Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation | journal = Biochemistry |volume = 42 |issue = 30 | pages = 9221–6 |year = 2003 |pmid = 12885257 | doi = 10.1021/bi034593c}}</ref> С друге стране, оксиданси попут [[Radikal (hemija)|слободних радикала]] или [[водоник пероксид]]а производе вишеструке форме оштећења, као што су модификације база, посебно гуанозина, и прекиди двоструких ланаца.<ref>{{cite journal |author = Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S |title=Hydroxyl radicals and DNA base damage | journal = Mutat Res |volume = 424 |issue = 1–2 | pages = 9–21 |year = 1999 |pmid = 10064846 |doi = 10.1016/S0027-5107(99)00004-4}}</ref> Типична људска ћелија садржи око 150,000 база које су подлегле оксидативним оштећењима.<ref>{{cite journal |author = Beckman KB, Ames BN |title = Oxidative decay of DNA |journal = J. Biol. Chem. |volume = 272 |issue = 32 |pages = 19633–6 |year = 1997 |pmid = 9289489 |doi = 10.1074/jbc.272.32.19633}}</ref> Међу тим оксидативним озледама, најопаснији су прекиди двоструких ланаца, јер се они тешко поправљају и могу да произведу [[генске мутације]], [[Umetanje (genetika)|генетичка уметања]] и [[Delecija|делеције]] из ДНК секвенце, као и [[Hromozomska translokacija|хромозомске транслокације]].<ref>{{cite journal |author = Valerie K, Povirk L |title = Regulation and mechanisms of mammalian double-strand break repair | journal = Oncogene |volume = 22 |issue = 37 | pages = 5792–812 |year = 2003 |pmid = 12947387 | doi = 10.1038/sj.onc.1206679}}</ref> |
ДНК може да буде оштећена многим врстама [[мутаген]]а, који мењању ДНК секвенцу. Мутагени обухватају [[Oksidacioni angens|оксидационе агенсе]],<ref>{{cite journal |title = DNA damage profiles induced by oxidizing agents |author = B. Epe |journal = Reviews of Physiology Biochemistry and Pharmacology |volume = 127 |year = 1996 |volume = 127 |pages = 223-249 |doi = 10.1007/BFb0048268 |url = http://www.springerlink.com/content/l4q086753q55765m/}}</ref><ref>{{cite journal |title = Quantitation of 8-Oxoguanine and Strand Breaks Produced by Four Oxidizing Agents |author = Laura J. Kennedy, Kenneth Moore Jr., Jennifer L. Caulfield, Steven R. Tannenbaum, and Peter C. Dedon |journal = Chem. Res. Toxicol. |year = 1997 |volume = 10 |issue = 4 |pages = 386–392 |doi = 10.1021/tx960102w |url = http://pubs.acs.org/doi/abs/10.1021/tx960102w}}</ref><ref>{{cite journal |title = Lipid peroxidation—DNAdamage by malondialdehyde |author = Lawrence J. MarnettCorresponding author contact information |journal = Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis |volume = 424 |issues = 1–2 |year = 1999 |pages = 83–95 |url = http://www.sciencedirect.com/science/article/pii/S002751079900010X}}</ref> [[Alkilacija|алкилирајуће агенсе]],<ref>{{cite journal |title = Alkylating DNA damage stimulates a regulated form of necrotic cell death |author = Wei-Xing Zong1, Dara Ditsworth, Daniel E. Bauer, Zhao-Qi Wang, and Craig B. Thompson |doi = 10.1101/gad.1199904 |journal = Genes & Dev. |year = 2004 |volume = 18 |pages = 1272-1282 |url = http://genesdev.cshlp.org/content/18/11/1272.short}}</ref><ref>{{cite journal |title = High-dose combination alkylatingagents with autologous bone-marrow support in patients with breast cancer: preliminary assessment of DNAdamage in individual peripheral blood lymphocytes using the single cell gel electrophoresis assay |author = Raymond R., Gary H.S. Straussb, William P. Peters |journal = Mutation Research/Environmental Mutagenesis and Related Subjects |volume = 271 |issue = 2 |year = 1992 |pages = 101–113 |url = http://www.sciencedirect.com/science/article/pii/0165116192910834}}</ref> као и [[Електромагнетско зрачење|електромагнетну радијацију]] високе енергије, попут [[Ultraljubičasto zračenje|ултраљубичастог]] светла<ref>{{cite journal |title = UV Light as a Footprinting Agent: Modulation of UV-induced DNADamage by Transcription Factors Bound at the Promoters of Three Human Genes |author = Silvia Tornaletti, Gerd P. Pfeifer |journal = Journal of Molecular Biology |volume = 249 |issue = 4 |year = 1995 |pages = 714–728 |url = http://www.sciencedirect.com/science/article/pii/S0022283685703312}}</ref><ref>{{cite journal |title = Wavelength dependence of oxidative DNA damage induced by UV and visible light |author = C Kielbassa, L Roza and B Epe |journal = Carcinogenesis |year = 1997 |volume = 18 |issue = 4 |pages = 811-816 |doi = 10.1093/carcin/18.4.811 |url = http://carcin.oxfordjournals.org/content/18/4/811.short}}</ref> и [[Rendgenski zraci|X-зрака]].<ref>{{cite journal |title = Evidence for a lack of DNA double-strand break repair in human cells exposed to very low x-ray doses |author = Kai Rothkamm and Markus Löbrich |doi = 10.1073/pnas.0830918100 |journal = PNAS |year = 2003 |volume = 100 |issue = 9 |pages = 5057-5062 |url = http://www.pnas.org/content/100/9/5057.short}}</ref><ref>{{cite journal |title = Enhancement of Xray induced DNAdamage by pre-treatment with halogenated pyrimidine analogs |author = Timothy J. Kinsella, Patricia P. Dobson, James B. Mitchell, Albert J. Fornace Jr. |journal = International Journal of Radiation |volume = 13 |issue = 5 |year = 1987 |pages = 733–739 |url = http://www.sciencedirect.com/science/article/pii/0360301687902926}}</ref> Тип произведеног ДНК оштећења зависи од типа мутагена. На пример, УВ светло може да оштети ДНК формирањем [[Pirimidinski dimer|тиминских димера]], који су међусобно повезани између пиримидинских база.<ref>{{cite journal |author = Douki T, Reynaud-Angelin A, Cadet J, Sage E |title = Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation | journal = Biochemistry |volume = 42 |issue = 30 | pages = 9221–6 |year = 2003 |pmid = 12885257 | doi = 10.1021/bi034593c}}</ref> С друге стране, оксиданси попут [[Radikal (hemija)|слободних радикала]]<ref>{{cite journal |title = Oxyradicals and DNA damage |author = Lawrence J. Marnett |journal = Carcinogenesis |year = 2000 |volume = 21 |issue = 3 |pages = 361-370 |doi= 10.1093/carcin/21.3.361 |url = http://carcin.oxfordjournals.org/content/21/3/361.short}}</ref><ref>{{cite journal |title = Enzymatic Processing of Radiation-Induced Free Radical Damage in DNA |author = Susan S. Wallace |journal = Radiation Research |year = 1998 |volume = 150 |issue = 5s |pages = S60-S79 |url = http://www.rrjournal.org/doi/abs/10.2307/3579809}}</ref> или [[водоник пероксид]]а<ref>{{cite journal |title = Comparison of the effects of hydrogen peroxide and X-ray irradiation on toxicity, mutation, and DNAdamage/repair in mammalian cells (V-79) |author = Matthews O Bradleya, Leonard C Erickson |journal = Biochimica et Biophysica Acta (BBA) - Nucleic Acids and Protein Synthesis |volume = 654 |issue = 1 |year = 1981 |pages = 135–141 |url = http://www.sciencedirect.com/science/article/pii/0005278781901465}}</ref><ref>{{cite journal |title = Toxic DNA damage by hydrogen peroxide through the Fenton reaction in vivo and in vitro |author = JA Imlay, SM Chin and S Linn |journal = Science |year = 1988 |volume = 240 |issue = 4852 |pages = 640-642 |doi = 10.1126/science.2834821 |url = http://www.sciencemag.org/content/240/4852/640.short}}</ref> производе вишеструке форме оштећења, као што су модификације база, посебно гуанозина, и прекиди двоструких ланаца.<ref>{{cite journal |author = Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S |title=Hydroxyl radicals and DNA base damage | journal = Mutat Res |volume = 424 |issue = 1–2 | pages = 9–21 |year = 1999 |pmid = 10064846 |doi = 10.1016/S0027-5107(99)00004-4}}</ref> Типична људска ћелија садржи око 150,000 база које су подлегле оксидативним оштећењима.<ref>{{cite journal |author = Beckman KB, Ames BN |title = Oxidative decay of DNA |journal = J. Biol. Chem. |volume = 272 |issue = 32 |pages = 19633–6 |year = 1997 |pmid = 9289489 |doi = 10.1074/jbc.272.32.19633}}</ref> Међу тим оксидативним озледама, најопаснији су прекиди двоструких ланаца, јер се они тешко поправљају и могу да произведу [[генске мутације]], [[Umetanje (genetika)|генетичка уметања]] и [[Delecija|делеције]] из ДНК секвенце, као и [[Hromozomska translokacija|хромозомске транслокације]].<ref>{{cite journal |author = Valerie K, Povirk L |title = Regulation and mechanisms of mammalian double-strand break repair | journal = Oncogene |volume = 22 |issue = 37 | pages = 5792–812 |year = 2003 |pmid = 12947387 | doi = 10.1038/sj.onc.1206679}}</ref> |
||
Многи мутагени се уклапају у простор између два суседна базна пара. То се назива [[interkalacija (hemija)|''интеркалација'']]. Већина интеркалатора су [[aromatičnost|ароматични]] и планарни молекули. Примери су [[етидијум бромид]], [[акридин]]и, [[Daunorubicin|дауномицин]], и [[доксорубицин]]. Да би интеркалатор могао да се уклопи између пара база, оне се морају раздвојити. Стога долази до дисторзије ДНК ланаца путем одвијања двоструког хеликса. Тиме се инхибирају транскрипција и репликација ДНК, што узрокује токсичност и мутације.<ref>{{cite journal |author = Ferguson L, Denny W |title = The genetic toxicology of acridines | journal = Mutat Res |volume = 258 |issue = 2 | pages = 123–60 |year = 1991 |pmid = 1881402}}</ref> Резултат је да ДНК интеркалатори могу да буду [[карциноген]]и, и у случају [[талидомид]]а, [[тератоген]]и.<ref>{{cite journal |author = Stephens T, Bunde C, Fillmore B |title = Mechanism of action in thalidomide teratogenesis |journal = Biochem Pharmacol |volume = 59 |issue = 12 |pages = 1489–99 |year = 2000 |pmid = 10799645 |doi = 10.1016/S0006-2952(99)00388-3}}</ref> Други попут [[benzo(a)pyren|бензо[''а'']пирен диол епоксида]] и [[афлатоксин]]а формирају ДНК адукте који индукују грешке у репликацији.<ref>{{cite journal |author = Jeffrey A |title = DNA modification by chemical carcinogens | journal = Pharmacol Ther |volume = 28 |issue = 2 | pages = 237–72 |year = 1985 |pmid = 3936066 |doi = 10.1016/0163-7258(85)90013-0}}</ref> Упркос томе, услед њихове способности да инхибирају ДНК транскрипцију и репликацију, други слични токсини се такође користе у [[hemoterapija|хемотерапији]] за инхибирање брзог раста ћелија [[канцер]]а.<ref>{{cite journal |author = Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A |title = Intercalators as anticancer drugs | journal = Curr Pharm Des |volume = 7 |issue = 17 | pages = 1745–80 |year = 2001 |pmid = 11562309 |doi = 10.2174/1381612013397113}}</ref> |
Многи мутагени се уклапају у простор између два суседна базна пара. То се назива [[interkalacija (hemija)|''интеркалација'']]. Већина интеркалатора су [[aromatičnost|ароматични]] и планарни молекули. Примери су [[етидијум бромид]], [[акридин]]и, [[Daunorubicin|дауномицин]], и [[доксорубицин]]. Да би интеркалатор могао да се уклопи између пара база, оне се морају раздвојити. Стога долази до дисторзије ДНК ланаца путем одвијања двоструког хеликса. Тиме се инхибирају транскрипција и репликација ДНК, што узрокује токсичност и мутације.<ref>{{cite journal |author = Ferguson L, Denny W |title = The genetic toxicology of acridines | journal = Mutat Res |volume = 258 |issue = 2 | pages = 123–60 |year = 1991 |pmid = 1881402}}</ref> Резултат је да ДНК интеркалатори могу да буду [[карциноген]]и, и у случају [[талидомид]]а, [[тератоген]]и.<ref>{{cite journal |author = Stephens T, Bunde C, Fillmore B |title = Mechanism of action in thalidomide teratogenesis |journal = Biochem Pharmacol |volume = 59 |issue = 12 |pages = 1489–99 |year = 2000 |pmid = 10799645 |doi = 10.1016/S0006-2952(99)00388-3}}</ref><ref>{{cite journal |title= Free radical-mediated oxidative DNA damage in the mechanism of thalidomide teratogenicity |author = Toufan Parman, Michael J. Wiley & Peter G. Wells |journal = Nature Medicine |volume = 5 |pages = 582 - 585 |year = 1999 |doi = 10.1038/8466 |url = http://www.nature.com/nm/journal/v5/n5/abs/nm0599_582.html}}</ref> Други попут [[benzo(a)pyren|бензо[''а'']пирен диол епоксида]] и [[афлатоксин]]а формирају ДНК адукте који индукују грешке у репликацији.<ref>{{cite journal |author = Jeffrey A |title = DNA modification by chemical carcinogens | journal = Pharmacol Ther |volume = 28 |issue = 2 | pages = 237–72 |year = 1985 |pmid = 3936066 |doi = 10.1016/0163-7258(85)90013-0}}</ref> Упркос томе, услед њихове способности да инхибирају ДНК транскрипцију и репликацију, други слични токсини се такође користе у [[hemoterapija|хемотерапији]] за инхибирање брзог раста ћелија [[канцер]]а.<ref>{{cite journal |author = Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A |title = Intercalators as anticancer drugs | journal = Curr Pharm Des |volume = 7 |issue = 17 | pages = 1745–80 |year = 2001 |pmid = 11562309 |doi = 10.2174/1381612013397113}}</ref> |
||
== Биолошка функција == |
== Биолошка функција == |
||
Верзија на датум 20. април 2012. у 17:02

Дезоксирибонуклеинска киселина (скраћено ДНК) је нуклеинска киселина која садржи упутства за развој и правилно функционисање свих живих организама. Заједно са РНК и протеинима, ДНК је један од три главна макромолекула који су есенцијални за све познате форме живота. Сва жива бића свој генетички материјал носе у облику ДНК, са изузетком неких вируса који имају РНК (рибонуклеинска киселина). ДНК има веома важну улогу не само у преносу генетичких информација са једне на другу генерацију, већ садржи упутства и за грађење неопходних ћелијских органела, протеина и РНК молекула. ДНК сегмент који преноси ова важна упуства се назива ген.[1]
У еукариотима, организмима као што су животиње, биљке, гљиве и протисте, највећи део ДНК молекула се налази у нуклеусу ћелије, а мањи део је у органелама, као што су митохондрије или хлоропласти.[2] У прокариотима (нпр. бактеријама) ДНК се налази у цитоплазми ћелије. За разлику од ензима, ДНК молекул не утиче директно на друге молекуле, већ различити ензими сарађују са ДНК и копирају информације било у облику дуплог ДНК молекула или у облику протеина. Овакав однос део је централне догме молекуларне биологије.[3]
Ћелије садрже ДНК организован у дуге структуре које се зову хромозоми. Током ћелијске деобе хромозоми се дуплирају процесом репликације ДНК, тако да свака од новонасталих ћелија има комплетан сет хромозома. У хромозомима хроматински протеини као што су хистони организују ДНК на такав начин да молекул постаје веома компактан и може да стане у ћелије које су на хиљаде пута мање од ДНК молекула. Ове компактне структуре условаљавају интеракције између ДНК и других протеина, и помажу у контролисању делова ДНК који се транскрибују.[4]
ДНК је дугачак полимер састављен од мањих јединица које се називају нуклеотиди. ДНК се састоји од два полимерна ланца који имају антипаралелну оријентацију. Међусобно повезани нуклеотиди чине скелетон ДНК молекула формираног од шећера и фосфатних група. Овај скелетон такође садржи четири различите нуклеобазе везане за моносахаридне јединице. Редослед ове четири базе је основа кодирања генетичког материјала. Информација се чита користећи генетички код, којим се специфицира секвенца аминокиселина у протеинима. Код се чита копирањем делова ДНК у сродне РНК молекуле у процесу који се назива транскрипција.
Особине


ДНК молекул је дугачак полимер који је састављен од нуклеотида, јединица које се понављају.[5][6][7] Нуклеотиди су веома мале јединице, међутим ДНК молекуле се састоји од милиона нуклеотида, што чини овај молекул веома дугим. Највећи људски хромозом се састоји од 440 милиона нуклеотида, односно 220 милиона пари.[8] База која је повезана са шећером назива се нуклеозид, док база која је повезана са шећером и једном или више фосфатних група се назива нуклеотид.[9] Када је више нуклеотида међусобно повезано, као нпр. у ДНК молекулу, тај полимер се онда назива полинуклеотидни ланац.[10]
Вотсон и Крик су 1953. показали да је у живим организмима ДНК молекул састављен од два полинуклеотидна ланца који су спирално увијени један око другоги, и за то откриће добили Нобелову награду. Вертикална дужина сваког обртаја спирале је 34 Ангстрема (3,4 nm) и пречник је 10 Ангстрема (1,0 nm).[11][12] Према једној другој студији, када се мерење изврши у одређеном раствору ДНК ланац је 22 до 26 Ангстрома широк (2,2 до 2,6 nm), и једна нуклеотидна јединица доприноси дужини са 3,3 Å (0,33 nm).[13]
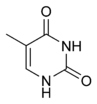
Шећер у ДНК молекулу је пентоза (назван тако јер садржи пет угљеникових атома) 2-дезоксирибоза (РНК молекул се састоји од шећера рибозе, отуда и пун назив рибонуклеинска киселина). Шећери су међусобно повезани фосфатним групама које стварају фосфодиестарску везу између трећег и петог угљениковог атома шећерног прстена. Фосфодиестарске везе су асиметричне те ДНК полинуклеотидни ланц имају свој правац. Како ови ланци иду у супротном правцу, можемо да кажемо да је ДНК антипаралелна. Асиметрични крајеви ДНК база се означавају са 5' (пет прим) и 3' (три прим). Антипаралелност значи да један ланац може ићи у правцу 5'→ 3', док супротни ланац иде у правцу 3'→ 5'. Спирални ланац који чини ДНК се одржава у том облику помоћу водоничних веза међу паровима двеју база.[14] У ДНК молекулу постоје четири базе Аденин (А), Цитозин (Ц), Гуанин (Г) и Тимин (Т):[12]
Четири базе су међусобно комплементарне: аденин (А) једног ланца је увек у пару са тимином (Т) другог, наспрамног ланца, и повезан је двема водоничним везама (и обрнуто). Гуанин (Г) једног ланца је увек у пару са цитозином (Ц) наспрамног ланца (и обрнуто), и повезан је трима водоничним везама. Сваки пар база ротира у односу на суседни за 36°, тако да сваки обртај спирале два полинуклеотидна ланца чине десет парова база (А-Т и Г-Ц). Полинуклеотидни ланци ротирају у правцу супротном од казаљки на сату.
Базе су подељене у две групе - пурини (аденин и гуанин) и пиримидине (цитозин и тимин). Урацил (У) база која се налази у РНК молекулу и која замењује тимин, припада пиримидинској групи. Једина разлика између тимина и урацила је недостатак једне метил груп код урацила. Урацил је стандардна база РНК молекула, и не постоји као таква у ДНК молекулу. Једини, тренутно познати, изузетак је бактеријски вирус ПБС1 који у свом ДНК молекулу има урацил као саставну базу[15]. Осим РНК и ДНК велики број вештачких аналога нуклеинских киселине је креиран ради студирања особина нуклеинских киселина, као и за примену у биотехнологији.[16]
Жлебови

Два комплементарна хеликсна ланца формирају основу ДНК молекула. Препознатљив је и двоструки хеликс који прати отворе, или жлебове, између ланаца. Та удубљења су непосредно поред парова база, те могу да служе као места везивања протеина и малих молекула. Пошто ланци нису директно насупрот један другом, жлебови немају једнаку величину. Велики жлеб је 22 Å широк, док мали жлеб има 12 Å.[20] Постоји низ изузетака, првенствено у случајевима необичних ДНК конформација. Називи велики и мали жлеб се увек односе на разлике у величини удубљења кад је ДНК у обичној Б форми.
Базе малог жлеба су на подеснијем растојању за везивање лиганда, него базе великог жлеба. Из тог разлога се протеини попут траскрипционих фактора који се везују за специфичне секвенце двоструког ДНК хеликса обично формирају водоничне везе са изложеним странама база малог жлеба.[21]
Денатурација и хибридизација ДНК
Секундарна структура ДНК је подложна денатурацији. Под денатурацијом се подразумева нарушавање секундарне структуре тако да се дволанчани ДНК молекул раздваја на два полинуклеотидна ланца. Под одговарајућим условима може доћи до ренатурације, тј. до поновног спајања комплементарних ланаца ДНК.[22] Процеси денатурације и ренатурације одигравају се и у ћелији под контролисаним условима и у ограниченом обиму. Ти процеси представљају неопходан предуслов за нормално функционисање ДНК, односно за њену репликацију, транскрипцију па тиме и транслацију.
Када се у раствору нађу два полинуклеотидна ланца који имају комплементарне редоследе нуклеотида, наградиће се хибридни дволанчани молекул.[23][24] Денатурисана ДНК може да хибридизује са денатурисаном ДНК исте или различите врсте, или са РНК. Хибридизација је нашла веома широку примену у истраживањима у молекуларној биологији и представља једну од основних техника генетичког инжењеринга.
Спаривање база


|

|
Свака база једног ланца се спарује само са једном базом на насупрутном ланцу. Овакво спаривање се назива комплементарно спаривање. Пурин се спарује са пиримидином водоничним везама, те се А спарује само са Т (двема водоничним везама) и Ц само са Г (са три водоничне везе). Како водоничне везе нису ковалентне, оне се лако раскидају и лако поново формирају. Ове везе се раскидају или механичким силама (нпр. током репликације) или високом температуром[25]. Две водоничне везе се лакше раскидају од три. Овај податак је битан ако секвенца ДНК молекула није унапред позната. Кад je секвенца ДНК молекула непозната, у молекуларној биологији се између осталог примењује техника која користи температуру, то што је температура виша, то се ДНК молекул теже раскида, те се може претпостави да тај молекул ДНК има велики број Ц и Г база (и.е. висок ГЦ-садржај). ДНК са високим ГЦ-садржајем је стабилнија од ДНК са ниским ГЦ-садржајем.[26]
Директна последица нуклеотидне комплементарности је да су информације у дволанчаној секвенци ДНК хеликса дуплиране, што је витално за репликацију молекула. Ова реверзибилна и специфична интеракција између комплементарних парова база је критична за све функције ДНК у живим организмима.[6]
Као што је горе напоменуто, већина ДНК молекула се састоји од два полимерна ланца, спојена у хеликсну структуру нековалентним везама. Ова дволанчана структура (dsDNA) се у знатној мери одржава посредством интеракција међуланчаног слагања база, које се најјаче за Г,Ц стекове. Два ланца се могу раздвојити у процесу познатом као топљење, чиме се формирају два молекула (ssDNA). До топљења долази кад су услови подесни, као што су високе температуре, ниске концентрације соли и високе pH вредности (низак pH такође отапа ДНК, али пошто је ДНК нестабилна услед денатурације киселине, низак pH се ретко користи).
Стабилност dsDNA форме зависи не само од ГЦ-садржаја (% Г,Ц базних парова) него и од секвенце (пошто је формирање стекова зависно од ње), као и од дужине (дужи молекули су стабилнији). Стабилност се може мерити на различите начине. Уобичајен приступ је мерење температуре топљења (која се још назива Тм вредност), што је температура на којој се 50% дволанчаних молекула конвертује у једноланчане молекуле. Настали једноланчани ДНК молекули немају јединствен заједнички облик, мада су неке конформације стабилније од других.[27] Температура топљења је зависна од јонске снаге и ДНК концентрације. Консеквентно, ГЦ садржај и дужина двоструког ДНК ланца одређују јачину асоцијације између ланаца. Дуги ДНК хеликси са високим ГЦ садржајем имају ланце са јачим интеракцијама, док кратки хеликси са високим АТ садржајем имају ланце са слабијим интеракцијама.[28] У биологији, делови ДНК двоструких хеликса који се лако раздвајају, као што је ТАТААТ Прибнов-кутија у неким промотерима, теже да имају висок АТ садржај.[29]
Sense и antisense
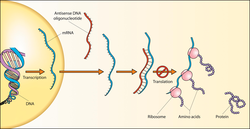
ДНК секвенца се назива sense (негативна (-)) ако је њена секвенца иста као и секвенца иРНК копије која се транслира у протеин.[30] Комплементарна секвенца супротног ланца се назива antisense (позитивна (+)') секвенца. Обе секвенце могу да постоје на различитим деловима истог ДНК ланца (и.е. оба ланца могу да садрже обе sense и antisense секвенце). Понекад се фраза кодирајући ланац среће. Међутим, кодирајућа и некодирајућа РНК могу да буду транскрибоване на сличан начин са оба ланца. У неким случајевима до транскрипције долази у оба правца почевши од заједничког промотерског региона, или до транскрипције може доћи унутар интрона, на оба ланца.[31][32][33]
Код прокариота и еукариота, antisense РНК секвенце се формирају, мада функција тих молекула није потпуно јасна.[34] Једна од претпоставки је да antisense РНК молекули учествују у регулацији експресије гена путем РНК-РНК спаривања база.[35]
Код малог броја ДНК секвенци прокариота у еукариота, и нешто већег броја код плазмида и вируса, разлика између sense и antisense ланаца је замагљена преклапањем гена.[36] У тим случајевима, неке ДНК секвенце има двоструку улогу. Оне кодирају један протеин кад се читају дуж једног ланца, и други протеин кад се читају у супротном правцу дуж другог ланца. Код бактерија, ово преклапање може да учествује у регулацији транскрипције ген,[37] док код вируса, преклапајући гени повећавају количину информације која може да буде кодирана унутар малог виралног генома.[38]
Супернамотавање


ДНК може да буде увијена попут канапа процесом који се назива ДНК супернамотавање. Кад је ДНК у свом „опуштеном“ стању, ланци обично обиђу осу двоструког хеликса једном свака 10,4 базна пара, док ако је ДНК упредена ланци постају више или мање збијени.[39] Ако је ДНК упредена у правцу хеликса, то је позитивно супернамотавање, и базе су ближе једна другој. Ако је ДНК упредена у супротном правцу, то је негативно супернамотавање, и базе се лакше растављају. У природи, ДНК је најчешће благо негативно супернамотана. То се остварује ензимимима који се зову топоизомеразе.[40] Ти ензими су исто тако потребни за отпуштање напрезања услед ДНК увијања насталог током процеса транскрипције и репликације ДНК.[41][42]
Хромозоми могу да буду веома велики, те средишњи сегменти могу да се понашају кад да су крајеви учвршћени. Резултат тога је да они не могу да расподеле сувишне намотаје на остатак хромозома, или да апсорбују завијање да би се опоравили од одвијања. Другим речима сегменти могу да постану супернамотани. У одговору на супернамотавање они ће бити изложени напрезању, као да су крајеви спојени.
DNK супернамотаванје је важно за DNA паковање унутар ћелија. Дужина ДНК је хиљадама пута већа од дужине ћелије, те је паковање генетичког материјала унутар ћелије или нуклеуса (код еукариота) комплексан задатак. Супернамотавање ДНК редукује потребни простор и омогућава паковање знатно веће количине ДНК. Код прокариота, плектонемски супернамотаји су предоминантни, зато што је хромозом кружног облика и садржи релативно малу количину генетичког материјала.[43][44][45][46][47] Код еукариота, ДНК супернамотавање се јавља на више нивоа плектонемских и соленоидалних супернамотаја, при чему се соленоидално супернамотавање показује најефективнијим у збијању ДНК молекула. Соленоидално супернамотавање се остварује путем хистона и формира се влакно са 10 nm у пресеку. Ово влакно се даље намотава у 30 nm широко влакно, које се даље намотава на само себе више пута.[48][49][50]
ДНК паковање се одвија у знатно повећаној мери током деобе једра у процесима митозе или мејозе, при којим се ДНК мора поделити и сажети у новонасталим ћелијама. Кондензини[51][52] и кохезини[53][54][55] су протеини за структурно одржавање хромозома који помажу у кондензацији хроматида и везивању њихових центромера. Ти протеини индукују позитивно супернамотавање.[56][57][58]
Алтернативне ДНК структуре



ДНК може да постоји у мноштву могућих конформација међу којима су А-ДНК[59], Б-ДНК[60][61][62], и З-ДНК форме[63][64], мада су једино Б-ДНК и З-ДНК директно примећене у функционалним организмима.[9] Конформација коју ДНК поприма зависи од нивоа хидратације, ДНК секвенце, количине и правца супернамотавања, хемијских модификација база, типа и концентрације металних јона, као и присуства полиамина у раствору.[65]
Први објављени извештаји о Рендгенској структури А-ДНК и Б-ДНК форми су користили анализу базирану на Патерсоновој трансформацији која даје само ограничену количину структурне информације о оријентацији ДНК влакана.[66][67][68] Један алтернативни аналитички приступ су предложили Вилкинс ет ал. 1953, за ин виво Б-ДНА дифракцију X-зрака/распореда максимума расејавања високо хидратисаних ДНК влакана базиран на квадратима Баселових функција.[69] У истом журналу, Џејмс Д. Ватсон и Францис Крик су објавили њихову анализу молекулског моделовања ДНК дифракционих образаца X-зрака и предложили структуру двоструког хеликса.[11]
Мада је Б-ДНК форма најчешћа под условима који владају у ћелијама,[70] она није добро дефинисана конформација него је фамилија сродних ДНК конформација[71] која се јавља при високим нивоима хидратације присутним у живим ћелијама. Њихови одговарајући рентгенски дифракциони обрасци расипања су карактеристични за молекулске паракристале са знатним степеном нереда.[72][73]
У поређењу са Б-ДНК, А-ДНА форма је шира деснорука спирала, са плитким, широким главним жлебом и ужим, дубљим малим жлебом. А форма се јавља под нефизиолошким условима у парцијално дехидратисаним ДНК узорцима, док се у ћелији може формирати при хибридном спаривању ДНК и РНК ланаца,[74][75][76] као и у ензим-ДНК комплексима.[77][78] Сегменти ДНК где су базе хемијски модификоване метилацијом могу да подлегну већим конформационим променама и да поприме З форму. Овде, ланци формирају леворуку спиралу око хеликсне осе, што је супротно уобичајеној Б форми.[79] Те необичне структуре се могу препознати по специфичним З-ДНК везујућим протеинима. Оне могу да учествују у регулацији транскрипције.[80][81][82][83][84][85]
Алтернативна ДНК хемија
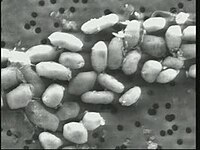

Током дужег низа година егзобиолози су предлагали постојање једне алтернативне биосфере која користи радикално различите биохемијске и молекулске процесе него тренутно познате животне форме. Једна од претпоставки је била постојање животног облика који користи арсеник уместо фосфора у ДНК.
На једној конференцији за штампу НАСА је децембра 2010 изјавила да батерија GFAJ-1, која је еволуирала у окружењу богатом у арсенику, прва земаљска животна форма која можда има ту способност.[86] Бактерија је нађена у Моно језеру, источно од Јесемитског националног парка. GFAJ-1 је штапићаста екстремофилна бактерија из фамилије Halomonadaceae, која у одсуству фосфора можда има способност инкорпорисања обично отровног елемента арсена у свој ДНК.[87] Ово откриће иде у прилог дугогодишњој идеји да би ванземаљски живот можда могао да има различиту биохемијску основу од живота на Земљи.[87][88] Истраживања је извео тип предвођен Фелисом Волф-Симон, која је геомикробиолог и геобиохемичар на НАСА астробиолошком институту при Државном универзитету Аризоне.
Овај налаз је наишао на јак критицизам у научној заједници. Научници тврде да нема доказа да је арсеник заправо инкорпориран у биомолекуле.[88][89] Микробиолог Јохан Хајдер је критиковао презентоване резултате студије. Он је упутио на могуће грешке у мерењу као и на погрешну интерпретацију резултата студије. По њему је, у оригиналној публикацији аутора поменуто загађење узорака остацима фосфата, који су вероватно присутни у довољној количини за основно снабдевање бактерија.[90] Независно потврђивање овог до сад није било могуће.
Квадруплексне структуре




На крајевима линеарних хромозома су специјализовани региони ДНК који се називају теломери.[92][93][94] Главна функција тих региона је да се омогући ћелији да репликује крајеве хромозома користећи ензим теломеразу, пошто ензими који нормално репликују ДНК не могу да копирају екстремне 3′ крајеве хромозома.[95][96] Ти специјализовани хромозомски завршеци такође помажу у заштити ДНК крајева, и спречавају системе за поправку ДНК у ћелији да их третирају као оштећења која треба поправљати.[97][98][99] У људским ћелијама, теломери се обично сегменти једноланчане ДНК који се састоје од неколико хиљада понављања једноставне TTAGGG секвенце.[100][101][102]
Ове гуанином богате секвенце могу да стабилизују хромозомске крајеве формирањем структура свежњева јединица са четири базе, уместо уобичајених базних парова ДНК молекула. Овде, четири гуанинске базе формирају равну површину, и те равне четворобазне јединице се затим слажу једна на другу да формирају стабилне Г-квадруплексне структуре (Г-тетраде Г4-ДНК).[103][104] Ове структуре су стабилизоване водоничним везивањем између база и хелацијом металног јона у центру сваке четворобазне јединице.[105] Низ других структуре се може формирати, са централним сетом од четири базе које долазе било из једног ланца савијеног око база, или неколико различитих паралелних ланаца, при чему сваки доприноси једну базу централној структури.
Осим ових стекованих структура, теломери исто тако формирају структуре са великим петљама које се називају теломерне петље, или Т-петље. Овде се једноланчана ДНК склупча у већи круг стабилизованом протеинима који се везују за теломере.[106] На самом крају Т-петље, једноланчана теломерна ДНК је спојена са регионом дволанчане ДНК тако што теломерни ланац делом ремети структуру ДНК двоструког хеликса и базно се спарује са једним од два ланца. Ова троланчана структура се назива депласманска петља или Д-петља.[103]
Квадруплекси се јављају не само у теломерима, него и на другим локацијама. На пример, за протоонкоген c-myc је показано[107][108] да формира квадруплекс у нуклеазном хиперсензитивном региону[109][110], који је критичан за активност гена.[111] Након тог иницијалног открића, за многе друге гене је нађено да имају Г-квадруплексе у њиховим промотерским регионима.[112] Неки од њих су живински β-глобински ген[113][114][115], људска убиквитинска лигаза RFP2[116][117][118] и протоонкогени c-kit[119][120][121], bcl-2[122][123][124], VEGF[125][126][127], H-ras[128][129][130] и N-ras[131][132][133].
Идентификација и предвиђање секвенци које имају способност формирања квадруплекса је важан корак у разумевању њихове улоге. Генерално једноставни обрасци се користе за претрагу могућих квадруплекс формирајућих секвенци: d(G3+N1-7G3+N1-7G3+N1-7G3+), при чему је N база (укључујући гуанин).[134][135] Ово правило је нашло широку примену у онлајн алгоритмима.
Прегледи целокупног генома базирани на првилу налажења квадруплекса су идентификовали 376,000 могућих квадруплексних секвенци (PQS) у људском геному. Знатан број њих се вероватно не формира ин виво.[135]. Једна слична студија је идентификовала могуће Г-квадруплекс код прокариота[136]. Постоји више модела који објашњавају како квадруплекси могу да контролишу активност гена. Један модел показује да формрање Г-квадруплекса на или близо промотера блокира транскрипцију гена, и тиме га деактивира.[137][138][139] У једном другом моделу квадруплекс формиран на некодирајућем ДНК ланцу помаже у одржавању отворене конформације кодирајућег ДНК ланца, те поспешује експресију респективног гена.[140][141][142]
Један начин индуковања или стабилизовања Г-квадруплексних формација, је увођење молекула који се могу везати за Г-квадруплексне структуре.[143][144] Познати су бројни лиганди, мали молекули и протеини који имају ту способност. Знатан број протеина који се јавља у природи се селективно везује за Г-квадруплексе. Међу њима су хеликазе,[145][146][147] које су имплициране у Блумов[148][149] и Вернеров синдром[148][150], и протеин RAP1 из Saccharomyces cerevisiae.[151][152][153] Развијен је протеин са цинк прст доменом[154] који се назва Gq1,[155][156][157] као и специфична антитела.[158][159][160]
Познато је да се катјонски порфирини везују за Г-квадруплексе,[161][162][163] као и молекул теломестатин.[164][165][166]
Разграната ДНК
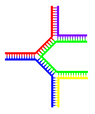
| |
| Једна грана | Вишеструко гранање |
До ДНК крзања долази кад некомплементарни региони постоје на једном или оба краја иначе комплементарне дволанчане ДНК. Разграната ДНК се може јавити ако се уведе трећи ДНК ланац који има способност хибридизације са отвореним ДНК сегментима дволанчане ДНК. Најједноставнији пример разгранавања је троланчана ДНК. Комплекси са додатним ланцима и вишеструким гранањем су такође познати.[168] Разграната ДНК налази примену у нанотехнологији.
Тест разгранате ДНК је тест амплификације сигнала (за разлику од теста амплификације биолошке мете) који се користи за детектовање молекула нуклеинских киселина.[169] Овај тест се може користити за детектовање и квантификацију многих типова РНК или ДНК. У тесту се разграната ДНК помеша са тестираним узорком. Детекција се врши користећи нерадиоактивни метод. Претходна амплификација нуклеинске киселине није неопходна. Тест је у потпуности завистан од хибридизације. Ензими се користе за одређивање степена хибридизације, али се не користе за манипулацију нуклеинских киселина. Мале количине нуклеинске киселине се могу детектовати и квантификовати без корака реверзне транскрипције (у случају РНК) и/или PCR[170][171]. Тест је подесан за употребу у високо проточном моду, за разлику од квантитативног Northern-blota[6][172] или теста РНК протекције.[173]
Вибрације
ДНК може да изводи ниско фреквентно колективно кретање. Оно се може мерити Рамановом спектроскопијом[174][175] и анализирати применом модела квази континуума.[176][177]
Модификације
Модификације база

|

|

|
| Цитозин | 5-метилцитозин | Тимин |
  |

Експресија гена је зависна од начина на који је ДНК пакована у хромозомима, у структурама званим хроматини. Модификације база могу да утичу на паковање. Региони који имају низак ниво или одсуство експресије обично садрже високе нивое метилације цитозинских база.[187] На пример, цитозинска метилација, производи 5-метилцитозин, који је важан за инактивацију X-хромозома.[188]
Просечни нивои метилације варирају између организама. Црв Caenorhabditis elegans не испољава цитозинску метилацију,[189][190] док кичмењаци имају високе нивое. До 1% њихове ДНК садржи 5-метилцитозин.[191]
ДНК метилација је кључни део нормалног развоја органисма и ћелијске диференцијације виших организама. ДНК метилација стабилно мења обрасце генског изражавања у ћелијама тако да оне могу да „запамте где су биле“ или да умање експресију гена. На пример, ћелије програмиране да буду Лангерхансова острвца током ембрионског развоја остају Лангерхансова острвца током животног века организма. ДНК метилација се типично уклања током формирања зигота и поново успоставља током накнадног ћелијског развоја. Недавна истраживања су показала да се метил групе заправо не уклањају у зиготима, него да долази до хидроксилације метил група.[192] Неке метилационе модификације које регулишу експресију гена су наследне и то се назива епигенетском регулацијом. ДНК метилација супресује изражавање виралних гена и других штетних елемената који су били инкорпорирани у геном домаћина током времена. ДНК метилација је исто тако основа хроматинске структуре, која омогућава ћелијама да поприме велики број карактеристика неопходних за мултицелуларни живот полазећи од једне непроменљиве ДНК секвенце.
ДНА метилација у позицији 5 цитозина има специфичан ефекат редуковања генске експресије и нађена је код свих кочмењака. У соматском ткиву одраслих особа, ДНК метилација се типично јавља у CpG динуклеотидном контексту, док је у ембрионским матичним ћелијама тренд супртоан.[193][194][195]
ДНК метилација је од пресудне важности у развоју скоро свих типова канцера.[196] Упркос важности 5-метилцитозина, може доћи до деаминације чиме се формира база тимин, тако да су метилисани цитозини посебно склони мутацијама.[197] Аберантни обрасци ДНК метилације су везани за велики број људских малигности и групишу се у две дистинктне форме: хиперметилација и хипометилација у односу на нормално ткиво. Хиперметилација је једна од главних епигенетских модификација које репресују транскрипцију путем промотерског региона тумор супресивних гена.[198][199][200] Хиперметилација се типично јавља на CpG острвима у промотерском региону те производи инактивацију гена. Глобална хипометилација је била имплицирана у развој и прогрес канцера путем различитих механизама.[201][202][203]
Модификације других база су метилација аденина код бактерија,[204][205] пресуство 5-хидроксиметилцитозина у мозгу,[206] и гликозилација урацила којом се формира "Ј-база" у кинетопластидима.[207][208]
Оштећења

ДНК може да буде оштећена многим врстама мутагена, који мењању ДНК секвенцу. Мутагени обухватају оксидационе агенсе,[212][213][214] алкилирајуће агенсе,[215][216] као и електромагнетну радијацију високе енергије, попут ултраљубичастог светла[217][218] и X-зрака.[219][220] Тип произведеног ДНК оштећења зависи од типа мутагена. На пример, УВ светло може да оштети ДНК формирањем тиминских димера, који су међусобно повезани између пиримидинских база.[221] С друге стране, оксиданси попут слободних радикала[222][223] или водоник пероксида[224][225] производе вишеструке форме оштећења, као што су модификације база, посебно гуанозина, и прекиди двоструких ланаца.[226] Типична људска ћелија садржи око 150,000 база које су подлегле оксидативним оштећењима.[227] Међу тим оксидативним озледама, најопаснији су прекиди двоструких ланаца, јер се они тешко поправљају и могу да произведу генске мутације, генетичка уметања и делеције из ДНК секвенце, као и хромозомске транслокације.[228]
Многи мутагени се уклапају у простор између два суседна базна пара. То се назива интеркалација. Већина интеркалатора су ароматични и планарни молекули. Примери су етидијум бромид, акридини, дауномицин, и доксорубицин. Да би интеркалатор могао да се уклопи између пара база, оне се морају раздвојити. Стога долази до дисторзије ДНК ланаца путем одвијања двоструког хеликса. Тиме се инхибирају транскрипција и репликација ДНК, што узрокује токсичност и мутације.[229] Резултат је да ДНК интеркалатори могу да буду карциногени, и у случају талидомида, тератогени.[230][231] Други попут бензо[а]пирен диол епоксида и афлатоксина формирају ДНК адукте који индукују грешке у репликацији.[232] Упркос томе, услед њихове способности да инхибирају ДНК транскрипцију и репликацију, други слични токсини се такође користе у хемотерапији за инхибирање брзог раста ћелија канцера.[233]
Биолошка функција
Паковање ДНК молекула у ћелијама
ДНК молекул је пристуан код еукариота и прокариота, и у оба случаја је саграђен од два спирално увијена полинуклеотидна ланца. Међутим, организација ДНК молекула код ових двеју организама је нешто другачија.
Готово код свих прокариота, ДНК молекул је затворен круг саграђен од два спирано увијена полинуклеотидна ланца. Код еукариота организација ДНК молекула је нешто компликованија. ДНК молекул је веома дугачак, у просеку до 2.5 метара. Та дужина молекула мора да стане у ћелије које су јако мало и не могу да се виде голим оком. Тако да ћелија мора да веома компактно спакује ДНК молекул, како би стао у тако сићушан простор. Начин на који је ово могуће је захваљујући протеинима који се зову хистони. Хистони су мали, основни протеини богати амино киселинама као што су лизин и аргинин. У еукариотским ћелијама је пронађено постојање пет хистона: H1, H2A, H2B, H3 и H4.
Хистони су у директном контакту са ДНК молекулом. Осам хистона (од сваког по два H2A, H2B, H3, H4), стварају структуре које изгледају као диск. Око сваког тог диска ДНК молекул се обавије, у дужини од 166 парова (А-Т и Ц-Г). Тако увијени ДНК молекул се обавије око јединог преосталог хистона H1, који не формира структуру у облику диска, већ служи само као веза до следећег диска (изграђеног од горепоменутих хистона) и поново се обавија око следећег диска.
Структура ДНК молекула обавијеног око диска се назива нуклеозом. Гледано кроз микроскоп свеукупна оваква структура изгледа као перлана огрлица. Овим непрекидним увијањем и савијањем ДНК молекула омогужава ћелији да стави ДНК молекул у свој веома мали простор.
Репликација ДНК молекула
Репликација ДНК молекула је веома сложен процес и веома важан процес. Цео живот, какав га ми знамо, заснива се на овом процесу. Због важности овог процеса, пуно времена, новца и труда је уложено на разумевање и откривање начина на којима се заснива репликација ДНК. У овом делу чланка биће описана репликација ДНК молекула код еукариота, а нешто касније и код прокариота.
Еукариотска репликација ДНК молекула
Репликација ДНК молекула почиње на месту који се зове oriC локус. Протеин ДНК-А се везује за oriC локус и при том се врши хидролиза АТП-а (аденозин-три-фосфат). Ово прво надовезивање доводи до почетног одвијања ДНК молекула из спирале у два линеарна ланца повезана водоничним везама. Да би репликација била успешна ДНК мора да постане линеаран, а не спирално увијен, дакле мора да изгледа као мердевине. Ензими који одвијају ДНК молекул у облик мердевине се зову хеликазе. Ензими одвијају ДНК молекул веома врзо, чак 75 до 100 револуција у секунди. Овакво веома брзо одвијање молекула ДНК може да доведе до стварања тензија полинуклеотидних ланаца. Ова појава тензија се може видети када се увију пертле и када покушамо брзо да их раздвојимо, пертле се увију у чвориће услед тензије. Да би се ово избегло, јер би овакво стварање чворова могло оштетити ДНК молекул, постоје ензими који се зову ДНК топоизомеразе, и који попуштају водоничне везе како би се тензија и стварање чворића избегло. У исто време док се ДНК молекул раздваја у облик мердевина, структура која се назива репликациона виљушка иде одмах иза топоизомераза и раздваја водоничне везе између парова (А-Т и Г-Ц). Да би ови полинуклеотидни ланци остали раздвојени раздвајајући протеини се везују на обе стране сваког ланца и на тај начин одржавају ланце одвојене. Репликација ДНК молекула може да се упореди са рајфелшусом. Када желимо да отворимо рајфелшус, вучемо механизам на доле, и на тај начин добијамо две стране рајфелшуса за раздвојеним зупчаницима. На исти начин се ДНК раздваја, где механизам на рајфелшусу представља репликациону виљушку.

Након раздвајања постоје два полинуклеотидна ланца, један иде у правцу 3'→ 5' док други иде у правцу 5'→ 3' (антипаралелност). Веома важан ензим који синтетише нове полинуклеотидне ланце ДНК Полимезара δ, може да синтетише нови ланац само у правцу 5'→ 3' читајући у правцу 3'→ 5'. То није проблем за водећи ланац који се синтетише у правцу кретања репликационе виљушке.
Синтезу оба ланца обавља ДНК полимераза тек пошто се веже за родитељски ланац који служи као матрица. Овај ензим не може да се веже за огољени ланац-матрицу већ захтева постојање зачетника (прајмера). Зачетник је кратки ланац РНК (на слици је представњен зеленом бојом)и његову синтезу катализује ензим примаза. Када се кратки ланац РНК комплементарно спари (хибридизује) са почетком ланца матрице то омогућује везивање ДНК полимеразе и почиње синтеза новог ланца. За синтезу ланца који заостаје потребно је да се синтетише већи број зачетника. Оказакијеве фрагменте, по завршетку синтезе, међусобно повезује ензим лигаза.
Ланац који се синтетише правцу супротном од правца кретања репликационе виљушке 3' → 5' није у могућности да буде комплетно синтетисан, он се синтетише у фрагментима који се називају Оказакијеви Фрагменти (названим по научнику Реији Оказаки који је први указао на њихово постојање) и појављују се само на ланцу који иде у овом правцу. Да би нови ДНК молекул био комплетан и без прекида, ензим лигаза има улогу лепка и везује фрагменте један за други, и тако добијемо од једног ДНК молекула, два новонастала ДНК молекула. ДНК Полимераза β има важну улогу у провери нових ДНК молекула, тако што иде дуж целих новонасталих ланаца и чита, и тиме проверава да су све базе повезане како треба да буду (А-Т и Г-Ц). ДНК репликација се зауставља када репликациона виљушка наиђе на секвенцу на ДНК молекулу који кодира за стопирање ДНК репликације.
Процес ДНК репликације је веома компликован, и један од разлога за ову комплексност је тај да новонастали ДНК молекули морају да буду тачни. Грешке у синтези ДНК молекула могу да доводе до разних болести и често су фаталне. Овај процес звучи веома невероватан када се узме у обзир да се нових 850 базних парова код прокариота синтетише у року од једне секунде, док код еукариота ова брзина је нешто нижа услед велике количине информације 150 базних парова у једној секунди.
Референце
- ^ Benjamin Lewin (1997). Genes (6th ed. изд.). Oxford University Press. ISBN 0198577796.
- ^ Russell, Peter (2001). iGenetics. New York: Benjamin Cummings. ISBN 0-8053-4553-1.
- ^ Brown T.A. (2006). Genomes (3rd ed. изд.). Garland Science. ISBN 0815341385.
- ^ Donald Voet; Judith G. Voet (2005). „DNA Replication, Repair, and Recombination”. Biochemistry (3 изд.). Wiley. ISBN 9780471193500.
- ^ Saenger Wolfram (1984). Principles of Nucleic Acid Structure. New York: Springer-Verlag. ISBN 0-387-90762-9.
- ^ а б в Bruce Alberts; Alexander Johnson; Julian Lewis; Martin Raff; Keith Roberts; Peter Walter (2002). Molecular Biology of the Cell. New York: Garlard Science. ISBN 0815332181.
- ^ Butler John M. (2001). Forensic DNA Typing. Elsevier. стр. 14—15. ISBN 978-0-12-147951-0. OCLC 223032110 45406517.
- ^ Gregory S, Barlow KF, McLay KE, Kaul R, Swarbreck D, Dunham A, Scott CE, Howe KL, Woodfine K (2006). „The DNA sequence and biological annotation of human chromosome 1”. Nature. 441 (7091): 315—21. Bibcode:2006Natur.441..315G. PMID 16710414. doi:10.1038/nature04727.
- ^ а б Ghosh A, Bansal M (2003). „A glossary of DNA structures from A to Z”. Acta Crystallogr D. 59 (4): 620—6. PMID 12657780. doi:10.1107/S0907444903003251.
- ^ Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents IUPAC-IUB Commission on Biochemical Nomenclature (CBN) Accessed 03 Jan 2006
- ^ а б Watson J.D. and Crick F.H.C. (1953). „A Structure for Deoxyribose Nucleic Acid” (PDF). Nature. 171 (4356): 737—738. Bibcode:1953Natur.171..737W. PMID 13054692. doi:10.1038/171737a0.
- ^ а б Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6
- ^ Mandelkern M, Elias J, Eden D, Crothers D (1981). „The dimensions of DNA in solution”. J Mol Biol. 152 (1): 153—61. PMID 7338906. doi:10.1016/0022-2836(81)90099-1.
- ^ Yakovchuk P, Protozanova E, Frank-Kamenetskii MD (2006). „Base-stacking and base-pairing contributions into thermal stability of the DNA double helix”. Nucleic Acids Res. 34 (2): 564—74. PMC 1360284
 . PMID 16449200. doi:10.1093/nar/gkj454.
. PMID 16449200. doi:10.1093/nar/gkj454.
- ^ Takahashi I, Marmur J. (1963). „Replacement of thymidylic acid by deoxyuridylic acid in the deoxyribonucleic acid of a transducing phage for Bacillus subtilis”. Nature. 197: 794—5. PMID 13980287.
- ^ Verma S, Eckstein F (1998). „Modified oligonucleotides: synthesis and strategy for users”. Annu. Rev. Biochem. 67: 99—134. PMID 9759484. doi:10.1146/annurev.biochem.67.1.99.
- ^ Mai-kun Teng, Nassim Usman, Christin A. Frederick and Andrew H.-J. Wang. „The molecular structure of the complex of Hoechst 33258 and the DNA dodecamer d(CGCGAATTCGCG)”. Nucl. Acids Res. 16 (6): 2671—2690. doi:10.1093/nar/16.6.2671. Непознати параметар
|yera=игнорисан (помоћ) - ^ Philip E. Pjura, Kazimierz Grzeskowiak, Richard E. Dickerson (1987). „Binding of Hoechst33258 to the minor groove of B-DNA”. Journal of Molecular Biology. 197 (2): 257—271. doi:10.1016/0022-2836(87)90123-9.
- ^ Frank G. Loontiens, Peter Regenfuss, Annelies Zechel, Lieve Dumortier, Robert M. Clegg (1990). „Binding characteristics of Hoechst 33258 with calf thymus DNA, poly[d(A-T)] and d(CCGGAATTCCGG): multiple stoichiometries and determination of tight binding with a wide spectrum of site affinities”. Biochemistry. 29 (38): 9029—9039. doi:10.1021/bi00490a021.
- ^ Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R (1980). „Crystal structure analysis of a complete turn of B-DNA”. Nature. 287 (5784): 755—8. Bibcode:1980Natur.287..755W. PMID 7432492. doi:10.1038/287755a0.
- ^ Pabo C, Sauer R (1984). „Protein-DNA recognition”. Annu Rev Biochem. 53: 293—321. PMID 6236744. doi:10.1146/annurev.bi.53.070184.001453.
- ^ C.G. Sibley and J.E. Ahlquist (1984). „The Phylogeny of the Hominoid Primates, as Indicated by DNA-DNA Hybridization”. Journal of Molecular Evolution. 20 (1): 2—15. PMID 6429338. doi:10.1007/BF02101980.
- ^ R.M. Myers, T. Maniatis, and L.S. Lerman (1987). „Detection and Localization of Single Base Changes by Denaturing Gradient Gel Electrophoresis”. Methods in Enzymology. 155: 501—527. ISBN 978-0-12-182056-5. PMID 3431470. doi:10.1016/0076-6879(87)55033-9.
- ^ T. Po, G. Steger, V. Rosenbaum, J. Kaper, and D. Riesner (1987). „Double-stranded cucumovirus associated RNA 5: experimental analysis of necrogenic and non-necrogenic variants by temperature-gradient gel electrophoresis”. Nucleic Acids Research. 15 (13): 5069—5083. PMC 305948
 . PMID 3601667. doi:10.1093/nar/15.13.5069.
. PMID 3601667. doi:10.1093/nar/15.13.5069.
- ^ Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H (2000). „Mechanical stability of single DNA molecules”. Biophys J. 78 (4): 1997—2007. PMID 10733978.
- ^ M. Mandel and J. Marmur (1968). „Use of Ultravialet Absorbance-Temperature Profile for Determining the Guanine plus Cytosine Content of DNA”. Methods in Enzymology. 12 (2): 198—206. ISBN 978-0-12-181856-2. doi:10.1016/0076-6879(67)12133-2.
- ^ Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J (2004). „Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern”. Biochemistry. 43 (51): 15996—6010. PMID 15609994. doi:10.1021/bi048221v.
- ^ Chalikian T, Völker J, Plum G, Breslauer K (1999). „A more unified picture for the thermodynamics of nucleic acid duplex melting: A characterization by calorimetric and volumetric techniques”. Proc Natl Acad Sci USA. 96 (14): 7853—8. Bibcode:1999PNAS...96.7853C. PMC 22151
 . PMID 10393911. doi:10.1073/pnas.96.14.7853.
. PMID 10393911. doi:10.1073/pnas.96.14.7853.
- ^ deHaseth P, Helmann J (1995). „Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA”. Mol Microbiol. 16 (5): 817—24. PMID 7476180. doi:10.1111/j.1365-2958.1995.tb02309.x.
- ^ Designation of the two strands of DNA JCBN/NC-IUB Newsletter 1989, Accessed 07 May 2008
- ^ Anne-Lise Haenni (2003). „Expression strategies of ambisense viruses”. Virus Research. 93 (2): 141—150.
- ^ Kakutani T, Hayano Y, Hayashi T, Minobe Y. (1991). „Ambisense segment 3 of rice stripe virus: the first instance of a virus containing two ambisense segments”. J Gen Virol. 72: 465—8.
- ^ Zhu Y, Hayakawa T, Toriyama S, Takahashi M. (1991). „Complete nucleotide sequence of RNA 3 of rice stripe virus: an ambisense coding strategy”. J Gen Virol. 72: 763—7.
- ^ Hüttenhofer A, Schattner P, Polacek N (2005). „Non-coding RNAs: hope or hype?”. Trends Genet. 21 (5): 289—97. PMID 15851066. doi:10.1016/j.tig.2005.03.007.
- ^ Munroe S (2004). „Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns”. J Cell Biochem. 93 (4): 664—71. PMID 15389973. doi:10.1002/jcb.20252.
- ^ Makalowska I, Lin C, Makalowski W (2005). „Overlapping genes in vertebrate genomes”. Comput Biol Chem. 29 (1): 1—12. PMID 15680581. doi:10.1016/j.compbiolchem.2004.12.006.
- ^ Johnson Z, Chisholm S (2004). „Properties of overlapping genes are conserved across microbial genomes”. Genome Res. 14 (11): 2268—72. PMC 525685
 . PMID 15520290. doi:10.1101/gr.2433104.
. PMID 15520290. doi:10.1101/gr.2433104.
- ^ Lamb R, Horvath C (1991). „Diversity of coding strategies in influenza viruses”. Trends Genet. 7 (8): 261—6. PMID 1771674. doi:10.1016/0168-9525(91)90326-L.
- ^ Benham C, Mielke S (2005). „DNA mechanics”. Annu Rev Biomed Eng. 7: 21—53. PMID 16004565. doi:10.1146/annurev.bioeng.6.062403.132016.
- ^ Champoux J (2001). „DNA topoisomerases: structure, function, and mechanism”. Annu Rev Biochem. 70: 369—413. PMID 11395412. doi:10.1146/annurev.biochem.70.1.369.
- ^ Wang J (2002). „Cellular roles of DNA topoisomerases: a molecular perspective”. Nat Rev Mol Cell Biol. 3 (6): 430—40. PMID 12042765. doi:10.1038/nrm831.
- ^ Albert A-C, Spirito F, Figueroa-Bossi N, Bossi L, Rahmouni AR (1996). „Hyper-negative template DNA supercoiling during transcription of the tetracycline-resistance gene in topA mutants is largely constrained in vivo”. Nucl Acids Res. 24 (15): 3093—3099. PMC 146055
 . PMID 8760899. doi:10.1093/nar/24.15.3093.
. PMID 8760899. doi:10.1093/nar/24.15.3093.
- ^ J. F. Marko, E. D. Siggia (1995). „Statistical mechanics of supercoiled DNA”. Phys. Rev. E. 52 (3): 2912—2938. doi:10.1103/PhysRevE.52.2912.
- ^ John F. Marko (1997). „Supercoiled and braided DNA under tension”. Phys. Rev. E. 55: 1758—1772. doi:10.1103/PhysRevE.55.1758.
- ^ A. Haller Igel, Manuel Ares Jr (1988). „Internal sequences that distinguish yeast from metazoan U 2 snRNA are unnecessary for pre-mRNA splicing” (PDF). Nature. 344 (4): 450—3.
- ^ John F. Marko (2007). „Torque and dynamics of linking number relaxation in stretched supercoiled DNA”. Phys. Rev. E. 76 (2). doi:10.1103/PhysRevE.76.021926.
- ^ T. Christian Boles Journal of Molecular Biology (1990). „Structure of plectonemically supercoiled DNA”. 213 (4): 931—951.
- ^ Victor F. Holmes and Nicholas R. Cozzarelli (2000). „Closing the ring: Links between SMC proteins and chromosome partitioning, condensation, and supercoiling”. PNAS. 97 (4): 1322—1324. doi:10.1073/pnas.040576797.
- ^ N.R. Cozzarelli, M.A. Krasnow, S.P. Gerrard, and J.H. White (1984). „A Topological Treatment of Recombination and Topoisomerases”. Cold Spring Harb Symp Quant Biol. 49: 383—400. doi:10.1101/SQB.1984.049.01.045.
- ^ H W Benjamin and N R Cozzarelli (1990). „Geometric arrangements of Tn3 resolvase sites”. The Journal of Biological Chemistry. 265: 6441—6447.
- ^ Hirano T (2005). „Condensins: organizing and segregating the genome”. Curr Biol. 15: R265—R275. PMID 15823530. doi:10.1016/j.cub.2005.03.037.
- ^ Wood AJ, Severson, AF, Meyer BJ (2010). „Condensin and cohesin complexity: the expanding repertoire of functions”. Nat Rev Genet. 11 (6): 391—404. PMID 20442714. doi:10.1038/nrg2794.
- ^ Michaelis C, Ciosk R, Nasmyth K (1997). „Cohesins: chromosomal proteins that prevent premature separation of sister chromatids”. Cell. 91 (1): 35—45. PMID 9335333. doi:10.1016/S0092-8674(01)80007-6. Непознати параметар
|month=игнорисан (помоћ) - ^ Guacci V, Koshland D, Strunnikov A (1997). „A Direct Link between Sister Chromatid Cohesion and Chromosome Condensation Revealed through the Analysis of MCD1 in S. cerevisiae”. Cell. 91 (1): 47—57. PMC 2670185
 . PMID 9335334. doi:10.1016/S0092-8674(01)80008-8. Непознати параметар
. PMID 9335334. doi:10.1016/S0092-8674(01)80008-8. Непознати параметар |month=игнорисан (помоћ) - ^ Tóth A, Ciosk R, Uhlmann F, Galova M, Schleiffer A, Nasmyth K (1999). „Yeast Cohesin complex requires a conserved protein, Eco1p(Ctf7), to establish cohesion between sister chromatids during DNA replication”. Genes Dev. 13 (3): 320—33. PMC 316435
 . PMID 9990856. doi:10.1101/gad.13.3.320. Непознати параметар
. PMID 9990856. doi:10.1101/gad.13.3.320. Непознати параметар |month=игнорисан (помоћ) - ^ Uhlmann F, Lottspeich F, Nasmyth K (1999). „Sister-chromatid separation at anaphase onset is promoted by cleavage of the cohesin subunit Scc1”. Nature. 400 (6739): 37—42. PMID 10403247. doi:10.1038/21831. Непознати параметар
|month=игнорисан (помоћ) - ^ Lee M. Silver (1995). „Karyotypes, Chromosomes, and Translocations”. Mouse Genetics: Concepts and Applications. Oxford: Oxford University Press. стр. 83—92. ISBN 978-0-19-507554-0.
- ^ Harvey Lodish, Arnold Berk, Chris A. Kaiser, Monty Krieger (2008). Molecular Cell Biology (6th изд.). New York: W.H. Freeman. ISBN 978-0-7167-7601-7.
- ^ Basham B, Schroth GP, Ho PS (1995). „An A-DNA triplet code: thermodynamic rules for predicting A- and B-DNA”. Proc Natl Acad Sci USA. 92 (14): 6464—6468. PMC 41538
 . PMID 7604014. doi:10.1073/pnas.92.14.6464.
. PMID 7604014. doi:10.1073/pnas.92.14.6464.
- ^ Dickerson RE (1989). „Definitions and nomenclature of nucleic acid structure components”. Nucleic Acids Res. 17 (5): 1797—1803. PMC 317523
 . PMID 2928107. doi:10.1093/nar/17.5.1797.
. PMID 2928107. doi:10.1093/nar/17.5.1797.
- ^ Lu XJ, Olson WK (1999). „Resolving the discrepancies among nucleic acid conformational analyses”. J Mol Biol. 285 (4): 1563—1575. PMID 9917397. doi:10.1006/jmbi.1998.2390.
- ^ Olson WK, Bansal M, Burley SK, Dickerson RE, Gerstein M, Harvey SC, Heinemann U, Lu XJ, Neidle S, Shakked Z, Sklenar H, Suzuki M, Tung CS, Westhof E, Wolberger C, Berman HM (2001). „A standard reference frame for the description of nucleic acid base-pair geometry”. J Mol Biol. 313 (1): 229—237. PMID 11601858. doi:10.1006/jmbi.2001.4987.
- ^ Wang AHJ, Quigley GJ, Kolpak FJ, Crawford JL, van Boom JH, Van der Marel G, Rich A (1979). „Molecular structure of a left-handed double helical DNA fragment at atomic resolution”. Nature (London). 282 (5740): 680—686. Bibcode:1979Natur.282..680W. PMID 514347. doi:10.1038/282680a0.
- ^ Ha SC, Lowenhaupt K, Rich A, Kim YG, Kim KK (2005). „Crystal structure of a junction between B-DNA and Z-DNA reveals two extruded bases”. Nature. 437 (7062): 1183—1186. Bibcode:2005Natur.437.1183H. PMID 16237447. doi:10.1038/nature04088.
- ^ Basu H, Feuerstein B, Zarling D, Shafer R, Marton L (1988). „Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies”. J Biomol Struct Dyn. 6 (2): 299—309. PMID 2482766.
- ^ Franklin RE, Gosling RG (6. 3. 1953). „The Structure of Sodium Thymonucleate Fibres I. The Influence of Water Content” (PDF). Acta Crystallogr. 6 (8–9): 673—7. doi:10.1107/S0365110X53001939.
- ^ Franklin RE, Gosling RG (1953). „The structure of sodium thymonucleate fibres. II. The cylindrically symmetrical Patterson function”. Acta Crystallogr. 6 (8–9): 678—85. doi:10.1107/S0365110X53001940.
- ^ Franklin, Rosalind and Gosling, Raymond (1953). „Molecular Configuration in Sodium Thymonucleate. Franklin R. and Gosling R.G” (PDF). Nature. 171 (4356): 740—1. Bibcode:1953Natur.171..740F. PMID 13054694. doi:10.1038/171740a0.
- ^ Wilkins M.H.F., A.R. Stokes A.R. & Wilson, H.R. (1953). „Molecular Structure of Deoxypentose Nucleic Acids” (PDF). Nature. 171 (4356): 738—740. Bibcode:1953Natur.171..738W. PMID 13054693. doi:10.1038/171738a0.
- ^ Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (1980). „Polymorphism of DNA double helices”. J. Mol. Biol. 143 (1): 49—72. PMID 7441761. doi:10.1016/0022-2836(80)90124-2.
- ^ Baianu, I.C. (1980). „Structural Order and Partial Disorder in Biological systems”. Bull. Math. Biol. 42 (4): 137—141. http://cogprints.org/3822/
- ^ Hosemann R., Bagchi R.N., Direct analysis of diffraction by matter, North-Holland Publs., Amsterdam – New York, 1962.
- ^ Baianu, I.C. (1978). „X-ray scattering by partially disordered membrane systems”. Acta Crystallogr A. 34 (5): 751—753. Bibcode:1978AcCrA..34..751B. doi:10.1107/S0567739478001540.
- ^ Igor Sidorenkov, Natalia Komissarova, Mikhail Kashle (1998). „Crucial Role of the RNA:DNAHybrid in the Processivity of Transcription”. Molecular Cell. 2 (1): 55—64.
- ^ G Milman, R Langridge, and M J Chamberlin (1967). „The structure of a DNA-RNA hybrid”. Proc Natl Acad Sci U S A. 57 (6): 1804—1810. Непознати параметар
|pmcid=игнорисан [|pmc=се препоручује] (помоћ) - ^ James Casey and Norman Davidson (1977). „Rates of formation and thermal stabilities of RNA:DNA and DNA:DNA duplexes at high concentrations of formamide”. Nucl. Acids Res. 4 (5): 1539—1552. doi:10.1093/nar/4.5.1539.
- ^ Wahl M, Sundaralingam M (1997). „Crystal structures of A-DNA duplexes”. Biopolymers. 44 (1): 45—63. PMID 9097733. doi:10.1002/(SICI)1097-0282(1997)44:1<45::AID-BIP4>3.0.CO;2-#.
- ^ Lu XJ, Shakked Z, Olson WK (2000). „A-form conformational motifs in ligand-bound DNA structures”. J. Mol. Biol. 300 (4): 819—40. PMID 10891271. doi:10.1006/jmbi.2000.3690.
- ^ Rothenburg S, Koch-Nolte F, Haag F (2001). „DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles”. Immunol Rev. 184: 286—98. PMID 12086319. doi:10.1034/j.1600-065x.2001.1840125.x.
- ^ Oh D, Kim Y, Rich A (2002). „Z-DNA-binding proteins can act as potent effectors of gene expression in vivo”. Proc. Natl. Acad. Sci. U.S.A. 99 (26): 16666—71. Bibcode:2002PNAS...9916666O. PMC 139201
 . PMID 12486233. doi:10.1073/pnas.262672699.
. PMID 12486233. doi:10.1073/pnas.262672699.
- ^ Louise H. Naylor and Elizabeth M. Clark (1990). „d(TG)n·d(CA)n sequences upstream of the rat prolactin gene form Z-DNA and inhibit gene transcription”. Nucl. Acids Res. 18 (6): 1595—1601. doi:10.1093/nar/18.6.1595.
- ^ Sharon Eden, Howard Cedar (1994). „Role of DNA methylation in the regulation of transcription”. Current Opinion in Genetics & Development. 4 (2): 255—259.
- ^ J.R. Broach, Y.-Y. Li, J. Feldman, M. Jayaram, J. Abraham, K.A. Nasmyth†, and J.B. Hicks (1983). „Localization and Sequence Analysis of Yeast Origins of DNA Replication”. Cold Spring Harb Symp Quant Biol. 47: 1165—1173. doi:10.1101/SQB.1983.047.01.132.
- ^ B Wittig, T Dorbic, and A Rich (1991). „Transcription is associated with Z-DNA formation in metabolically active permeabilized mammalian cell nuclei”. PNAS. 88 (6): 2259—2263.
- ^ S Wölfl, C Martinez, A Rich, and J A Majzoub (1996). „Transcription of the human corticotropin-releasing hormone gene in NPLC cells is correlated with Z-DNA formation”. PNAS. 93 (8): 3664—3668.
- ^ Wolfe-Simon Felisa, Blum Jodi Switzer, Kulp Thomas R., Gordon Gwyneth W., Hoeft Shelley E., Pett-Ridge Jennifer, Stolz John F., Webb Samuel M., Weber Peter K. (2. 12. 2010). „A bacterium that can grow by using [[arsenik|arsenic]] instead of phosphorus”. Science. 332 (6034): 1163—1166. PMID 21127214. doi:10.1126/science.1197258. Приступљено 9. 6. 2011. Сукоб URL—викивеза (помоћ)
- ^ а б „Arsenic-loving bacteria may help in hunt for alien life”. BBC News. 2. 12. 2010. Приступљено 2010-12-02.
|first1=захтева|last1=у Authors list (помоћ) - ^ а б Bortman, Henry (2010-12-02). „Arsenic-Eating Bacteria Opens New Possibilities for Alien Life”. Space.Com web site. Space.com. Приступљено 2010-12-02. Спољашња веза у
|work=(помоћ) - ^ Katsnelson, Alla (2. 12. 2010). „Arsenic-eating microbe may redefine chemistry of life”. Nature News. doi:10.1038/news.2010.645.
- ^ Kritik an Arsen-Bakterien-Fund, "Forschung aktuell"
- ^ NDB UD0017, Structure and packing of human telomeric DNK
- ^ Passarge, Eberhard (2006). Color atlas of genetics (3 Rev Upd изд.). Thieme. ISBN 1588903362.
- ^ Olovnikov Alexei M. (1971). „Принцип маргинотомии в матричном синтезе полинуклеотидов” [Principle of marginotomy in template synthesis of polynucleotides]. Doklady Akademii Nauk SSSR (на језику: руски). 201 (6): 1496—9. PMID 5158754.
- ^ Olovnikov AM (1973). „A theory of marginotomy. The incomplete copying of template margin in enzymic synthesis of polynucleotides and biological significance of the phenomenon”. J. Theor. Biol. 41 (1): 181—90. PMID 4754905. doi:10.1016/0022-5193(73)90198-7. Непознати параметар
|month=игнорисан (помоћ) - ^ Greider C, Blackburn E (1985). „Identification of a specific telomere terminal transferase activity in Tetrahymena extracts”. Cell. 43 (2 Pt 1): 405—13. PMID 3907856. doi:10.1016/0092-8674(85)90170-9.
- ^ „The Nobel Prize in Physiology or Medicine 2009”. The Nobel Foundation. 2009-10-05. Приступљено 2010-10-23.
- ^ Nugent C, Lundblad V (1998). „The telomerase reverse transcriptase: components and regulation”. Genes Dev. 12 (8): 1073—85. PMID 9553037. doi:10.1101/gad.12.8.1073.
- ^ Acharya PV (1971). „The isolation and partial characterization of age-correlated oligo-deoxyribo-ribonucleotides with covalently linked aspartyl-glutamyl polypeptides.”. Johns Hopkins medical journal. Supplement (1): 254—60. PMID 5055816.
- ^ Bjorksten J, Acharya PV, Ashman S, Wetlaufer DB (1971). „Gerogenic fractions in the tritiated rat.”. Journal of the American Geriatrics Society. 19 (7): 561—74. PMID 5106728.
- ^ Wright W, Tesmer V, Huffman K, Levene S, Shay J (1997). „Normal human chromosomes have long G-rich telomeric overhangs at one end”. Genes Dev. 11 (21): 2801—9. PMC 316649
 . PMID 9353250. doi:10.1101/gad.11.21.2801.
. PMID 9353250. doi:10.1101/gad.11.21.2801.
- ^ R K Moyzis, J M Buckingham, L S Cram, M Dani, L L Deaven, M D Jones, J Meyne, R L Ratliff, and J R Wu (1988). „A highly conserved repetitive DNA sequence, (TTAGGG)n, present at the telomeres of human chromosomes”. PNAS. 85 (18): 6622—6626.
- ^ Gregg B. Morin (1989). „The human telomere terminal transferase enzyme is a ribonucleoprotein that synthesizes TTAGGG repeats”. Cell. 59 (3): 521—529.
- ^ а б Burge S, Parkinson G, Hazel P, Todd A, Neidle S (2006). „Quadruplex DNA: sequence, topology and structure”. Nucleic Acids Res. 34 (19): 5402—15. PMC 1636468
 . PMID 17012276. doi:10.1093/nar/gkl655.
. PMID 17012276. doi:10.1093/nar/gkl655.
- ^ Neidle & Balasubramanian, ур. (2006). Quadruplex Nucleic Acids. ISBN 0-85404-374-8.
- ^ Parkinson G, Lee M, Neidle S (2002). „Crystal structure of parallel quadruplexes from human telomeric DNA”. Nature. 417 (6891): 876—80. PMID 12050675. doi:10.1038/nature755.
- ^ Griffith J, Comeau L, Rosenfield S, Stansel R, Bianchi A, Moss H, de Lange T (1999). „Mammalian telomeres end in a large duplex loop”. Cell. 97 (4): 503—14. PMID 10338214. doi:10.1016/S0092-8674(00)80760-6.
- ^ Simonsson, T.; Pecinka, P.; Kubista, M. (1998). „DNA tetraplex formation in the control region of c-myc”. Nucleic Acids Research. 26 (5): 1167—1172. PMC 147388
 . PMID 9469822. doi:10.1093/nar/26.5.1167.
. PMID 9469822. doi:10.1093/nar/26.5.1167.
- ^ Siddiqui-Jain, A.; Grand, C. L.; Bearss, D. J.; Hurley, L. H. (2002). „Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription”. Proceedings of the National Academy of Sciences. 99 (18): 11593—11598. PMC 129314
 . PMID 12195017. doi:10.1073/pnas.182256799.
. PMID 12195017. doi:10.1073/pnas.182256799.
- ^ D S Gross, and W T Garrard (1988). „Nuclease Hypersensitive Sites in Chromatin”. Annual Review of Biochemistry. 57: 159—197. doi:10.1146/annurev.bi.57.070188.001111.
- ^ A Almer and W Hörz (1986). „Nuclease hypersensitive regions with adjacent positioned nucleosomes mark the gene boundaries of the PHO5/PHO3 locus in yeast”. EMBO J. 5 (10): 2681—2687.
- ^ Johnson JE, Smith JS, Kozak ML, Johnson FB (2008). „In vivo veritas: using yeast to probe the biological functions of G-quadruplexes”. Biochimie. 90 (8): 1250—1263. PMC 2585026
 . PMID 18331848. doi:10.1016/j.biochi.2008.02.013.
. PMID 18331848. doi:10.1016/j.biochi.2008.02.013.
- ^ Xu Hou, Wei Guo, Fan Xia, Fu-Qiang Nie, Hua Dong, Ye Tian, Liping Wen, Lin Wang, Liuxuan Cao, Yang Yang, Jianming Xue, Yanlin Song, Yugang Wang, Dongsheng Liu, and Lei Jiang (2009). „A biomimetic potassium responsive nanochannel: G-quadruplex DNA conformational switching in a synthetic nanopore”. J. Am. Chem. Soc. 131 (22): 7800—7805. PMID 19435350. doi:10.1021/ja901574c.
- ^ Ren Zhang1, Yan Lin2 and Chun-Ting Zhang (2008). „Greglist: a database listing potential G-quadruplex regulated genes”. Nucl. Acids Res. 36 (suppl 1): D372—D376. doi:10.1093/nar/gkm787.
- ^ Phillip A. Rachwal, I. Stuart Findlow, Joern M. Werner, Tom Brown and Keith R. Fox (2007). „Intramolecular DNA quadruplexes with different arrangements of short and long loops”. Nucl. Acids Res. 35 (12): 4214—4222. doi:10.1093/nar/gkm316.
- ^ Antonio Randazzo, Veronica Esposito, Oliver Ohlenschläger, Ramadurai Ramachandran, Antonella Virgilio & Luciano Mayol (2005). „Structural studies on LNA quadruplexes”. Nucleosides, Nucleotides and Nucleic Acids. 24 (5-7): 795—800.
- ^ Mikhail Skoblova, Konstantin Shakhbazov, Dmitry Oshchepkov, Dmitry Ivanov, Anna Guskovaa, Dmitry Ivanov, Petr Rubtsov, Vladimir Prasolov, Nick Yankovsky, Ancha Baranova (2006). „Human RFP2 gene promoter: Unique structure and unusual strength”. Biochemical and Biophysical Research Communications. 342 (3): 859—866.
- ^ Aybike Birerdinc, Elizabeth Nohelty, Andrey Marakhonov, Ganiraju Manyam, Ivan Panov, Stephanie Coon, Eugene Nikitin, Mikhail Skoblov, Vikas Chandhoke and Ancha Baranova. „Pro-apoptotic and antiproliferative activity of human KCNRG, a putative tumor suppressor in 13q14 region”. Tumor Biology. 31 (1): 33—45. doi:10.1007/s13277-009-0005-0.
- ^ JianHua Gan, Jia Sheng and Zhen Huang. „Chemical and structural biology of nucleic acids and protein-nucleic acid complexes for novel drug discovery”. Science China Chemistry. 54 (1): 3—23. doi:10.1007/s11426-010-4174-x.
- ^ Kevin M. Sales, Marc C. Winslet and Alexander M. Seifalian. „Stem Cells and Cancer: An Overview”. Stem Cell Reviews and Reports. 3 (4): 249—255. doi:10.1007/s12015-007-9002-0.
- ^ Aintzane Apraiz, Maria Dolores Boyano email and Aintzane Asumendi (2011). „Cell-Centric View of Apoptosis and Apoptotic Cell Death-Inducing Antitumoral Strategies”. Cancers. 3 (1): 1042—1080. doi:10.3390/cancers3011042.
- ^ Stefan Duensinga, Anette Duensing (2010). „Targeted therapies of gastrointestinal stromal tumors (GIST)—The next frontiers”. Biochemical Pharmacology. 80 (5): 575—583.
- ^ Jixun Dai, Thomas S. Dexheimer, Ding Chen, Megan Carver, Attila Ambrus, Roger A. Jones, and Danzhou Yang (2006). „An Intramolecular G-Quadruplex Structure with Mixed Parallel/Antiparallel G-Strands Formed in the Human BCL-2 Promoter Region in Solution”. J. Am. Chem. Soc. 128 (4): 1096—1098. doi:10.1021/ja055636a.
- ^ Thomas S. Dexheimer, Daekyu Sun, and Laurence H. Hurley (2006). „Deconvoluting the Structural and Drug-Recognition Complexity of the G-Quadruplex-Forming Region Upstream of the bcl-2 P1 Promoter”. J. Am. Chem. Soc. 128 (16): 5404—5415. doi:10.1021/ja0563861.
- ^ Francesco Pezzella, Helen Turley, Isinzu Kuzu, Mohammed Fahim Tungekar, Michael S. Dunnill, Chris B. Pierce, Adrian Harris, Kevin C. Gatter, and David Y. Mason (1993). „bcl-2 Protein in Non-Small-Cell Lung Carcinoma”. N Engl J Med. 329: 690—694.
- ^ Daekyu Sun, Kexiao Guo, Jadrian J. Rusche and Laurence H. Hurley. „Facilitation of a structural transition in the polypurine/polypyrimidine tract within the proximal promoter region of the human VEGF gene by the presence of potassium and G-quadruplex-interactive agents”. Nucleic Acids Research. 33 (18): 6070—6080. doi:10.1093/nar/gki917.
- ^ Himesh Fernando, Anthony P. Reszka, Julian Huppert, Sylvain Ladame, Sarah Rankin, Ashok R. Venkitaraman, Stephen Neidle, and Shankar Balasubramanian (2006). „A Conserved Quadruplex Motif Located in a Transcription Activation Site of the Human c-kit Oncogene”. Biochemistry. 45 (25): 7854—7860. doi:10.1021/bi0601510.
- ^ Daekyu Sun, Wei-Jun Liu, Kexiao Guo, Jadrian J. Rusche, Scot Ebbinghaus, Vijay Gokhale and Laurence H. Hurley. „The proximal promoter region of the human vascular endothelial growth factor gene has a G-quadruplex structure that can be targeted by G-quadruplex–interactive agents”. Mol Cancer Ther April. 2008 (7): 880. doi:10.1158/1535-7163.MCT-07-2119.
- ^ Masood A. Shammas, Robert J. Shmookler Reis, Masaharu Akiyama, Hemanta Koley, Dharminder Chauhan, Teru Hideshima, Raj K. Goya, Laurence H. Hurley, Kenneth C. Anderson and Nikhil C. Munshi (2003). „Telomerase inhibition and cell growth arrest by G-quadruplex interactive agent in multiple myeloma”. Mol Cancer Ther. 2: 825.
- ^ Steven S. Smith, Ali Laayoun, Robert G. Lingeman, David J. Baker, Jezia Riley (1994). „Hypermethylation of Telomere-like Foldbacks at Codon 12 of the Human c-Ha-ras Gene and the Trinucleotide Repeat of the FMR-1 Gene of Fragile X”. Journal of Molecular Biology. 243 (2): 143—151.
- ^ Purnima R. Laud1, Asha S. Multani, Susan M. Bailey, Ling Wu, Jin Ma, Charles Kingsley, Michel Lebe, Sen Pathak, Ronald A. DePinho, and Sandy Chang (2005). „Elevated telomere-telomere recombination in WRN-deficient, telomere dysfunctional cells promotes escape from senescence and engagement of the ALT pathway”. Genes & Dev. 19: 2560—2570. doi:10.1101/gad.1321305.
- ^ Sunita Kumari, Anthony Bugaut, Julian L Huppert & Shankar Balasubramanian (2007). „An RNA G-quadruplex in the 5' UTR of the NRAS proto-oncogene modulates translation”. Nature Chemical Biology. 3: 218—221. doi:10.1038/nchembio864.
- ^ Mekala Gunaratnam, Stephen Swank, Shozeb M. Haider, Katja Galesa, Anthony P. Reszka, Monica Beltran, Francisco Cuenca, Jonathan A. Fletcher and Stephen Neidle (2009). „Targeting Human Gastrointestinal Stromal Tumor Cells with a Quadruplex-Binding Small Molecule”. J. Med. Chem. 52 (12): 3774—3783. doi:10.1021/jm900424a.
- ^ James E. Redman, J. M. Granadino-Roldán, James A. Schouten, Sylvain Ladame, Anthony P. Reszka, Stephen Neidle and Shankar Balasubramanian (2009). „http://pubs.acs.org/doi/abs/10.1021/jm900424a”. Org. Biomol. Chem. 7: 76—84. doi:10.1039/B814682A. Спољашња веза у
|title=(помоћ) - ^ Todd, A. K.; Johnston, M.; Neidle, S. (2005). „Highly prevalent putative quadruplex sequence motifs in human DNA”. Nucleic Acids Research. 33 (9): 2901—2907. PMC 1140077
 . PMID 15914666. doi:10.1093/nar/gki553.
. PMID 15914666. doi:10.1093/nar/gki553.
- ^ а б Huppert, J. L.; Balasubramanian, S. (2005). „Prevalence of quadruplexes in the human genome”. Nucleic Acids Research. 33 (9): 2908—2916. PMC 1140081
 . PMID 15914667. doi:10.1093/nar/gki609.
. PMID 15914667. doi:10.1093/nar/gki609.
- ^ Rawal, P.; Kummarasetti, V. B.; Ravindran, J.; Kumar, N.; Halder, K.; Sharma, R.; Mukerji, M.; Das, S. K.; et al. (2006). „Genome-wide prediction of G4 DNA as regulatory motifs: Role in Escherichia coli global regulation”. Genome Research. 16 (5): 644—655. PMC 1457047
 . PMID 16651665. doi:10.1101/gr.4508806.
. PMID 16651665. doi:10.1101/gr.4508806.
- ^ Susanna Cogoi and Luigi E. Xodo. „G-quadruplex formation within the promoter of the KRAS proto-oncogene and its effect on transcription”. Nucleic Acids Research. 34 (9): 2536—2549. doi:10.1093/nar/gkl286.
- ^ Thibault Lemarteleura, Dennis Gomeza, Rajaa Paterskia, Eliane Mandineb, Patrick Maillietb, Jean-François Riou (2004). „Stabilization of the c-mycgenepromoterquadruplex by specific ligands’ inhibitors of telomerase”. Biochemical and Biophysical Research Communications. 323 (3): 802—808.
- ^ Julian Leon Huppert (2008). „Four-stranded nucleic acids: structure, function and targeting of G-quadruplexes”. Chem. Soc. Rev. 37: 1375—1384. doi:10.1039/B702491F.
- ^ Attila Ambrus, Ding Chen, Jixun Dai, Tiffanie Bialis, Roger A. Jones and Danzhou Yang. „Human telomeric sequence forms a hybrid-type intramolecular G-quadruplex structure with mixed parallel/antiparallel strands in potassium solution”. Nucleic Acids Research. 34 (9): 2723—2735. doi:10.1093/nar/gkl348.
- ^ Yujian He, Ronald D. Neumann and Igor G. Panyutin. „Intramolecular quadruplex conformation of human telomeric DNA assessed with 125I-radioprobing”. Nucleic Acids Research. 32 (18): 5359—5367. doi:10.1093/nar/gkh875.
- ^ José P. Abad, Alfredo Villasante (1999). „The 3′ non-coding region of the Drosophila melanogaster HeT-A telomeric retrotransposon contains sequences with propensity to form G-quadruplexDNA”. FEBS letters. 453 (1–2): 59—62.
- ^ Richard H. Shafer (1997). „Stability and Structure of Model DNA Triplexes and Quadruplexes and Their Interactions with Small Ligands”. Progress in Nucleic Acid Research and Molecular Biology. 59: 55—94.
- ^ Tian-miao Ou, Yu-jing Lu, Jia-heng Tan, Zhi-shu Huang, Kwok-Yin Wong, Lian-quan Gu (2008). „G-Quadruplexes: Targets in Anticancer Drug Design”. 3: 690—713. doi:10.1002/cmdc.200700300. Непознати параметар
|isue=игнорисан (помоћ) - ^ Yuliang Wu, Kazuo Shin-ya and Robert M. Brosh Jr. (2008). „FANCJ Helicase Defective in Fanconia Anemia and Breast Cancer Unwinds G-Quadruplex DNA To Defend Genomic Stability”. Mol. Cell. Biol. 28 (12): 4116—4128. doi:10.1128/MCB.02210-07 Проверите вредност параметра
|doi=(помоћ). zero width space character у|doi=на позицији 9 (помоћ) - ^ Haiyong Han, Richard J. Bennett, and Laurence H. Hurley (2000). „Inhibition of Unwinding of G-Quadruplex Structures by Sgs1 Helicase in the Presence of N,N‘-Bis[2-(1-piperidino)ethyl]-3,4,9,10-perylenetetracarboxylic Diimide, a G-Quadruplex-Interactive Ligand”. Biochemistry. 39 (31): 9311—9316. doi:10.1021/bi000482r.
- ^ Haiyong Han, David R. Langley, Anupama Rangan, and Laurence H. Hurley (2001). „Selective Interactions of Cationic Porphyrins with G-Quadruplex Structures”. J. Am. Chem. Soc. 123 (37): 8902—8913. doi:10.1021/ja002179j.
- ^ а б James, William; Berger, Timothy; Elston, Dirk (2005). Andrews' Diseases of the Skin: Clinical Dermatology. (10th ed.). Saunders. ISBN 0-7216-2921-0.
- ^ Straughen, Je; Johnson, J; Mclaren, D; Proytcheva, M; Ellis, N; German, J; Groden, J (1998). „A rapid method for detecting the predominant Ashkenazi Jewish mutation in the Bloom's syndrome gene”. Human mutation. 11 (2): 175—8. PMID 9482582. doi:10.1002/(SICI)1098-1004(1998)11:2<175::AID-HUMU11>3.0.CO;2-W.
- ^ Ozgenc A, Loeb LA (2005). „Current advances in unraveling the function of the Werner syndrome protein”. Mutation research. 577 (1–2): 237—51. PMID 15946710. doi:10.1016/j.mrfmmm.2005.03.020. Непознати параметар
|month=игнорисан (помоћ) - ^ R Giraldo and D Rhodes (1994). „The yeast telomere-binding protein RAP1 binds to and promotes the formation of DNA quadruplexes in telomeric DNA”. EMBO J. 13 (10): 2411—2420. PMC 395107
 .
.
- ^ Steve G. Hershman, Qijun Chen, Julia Y. Lee, Marina L. Kozak, Peng Yue, Li-San Wang, and F. Brad Johnson (36). „Genomic distribution and functional analyses of potential G-quadruplex-forming sequences in Saccharomyces cerevisiae”. Nucl. Acids Res. (1): 144—156. doi:10.1093/nar/gkm986. Проверите вредност парамет(а)ра за датум:
|date=(помоћ) - ^ Peter König1, Rafael Giraldo, Lynda Chapman, Daniela Rhodes (1996). „The Crystal Structure of the DNA-Binding Domain of Yeast RAP1 in Complex with Telomeric DNA”. Cell. 85 (1): 125—136.
- ^ Miller J, McLachlan AD, Klug A (1985). „Repetitive zinc-binding domains in the protein transcription factor IIIA from Xenopus oocytes”. EMBO J. 4 (6): 1609—14. PMC 554390
 . PMID 4040853. Непознати параметар
. PMID 4040853. Непознати параметар |month=игнорисан (помоћ) - ^ Mark Isalan, Sachin D Patel, Shankar Balasubramanian, and Yen Choo (2001). „Selection of Zinc Fingers that Bind Single-Stranded Telomeric DNA in the G-Quadruplex Conformation”. 40 (3): 830—836. doi:10.1021/bi001728v.
- ^ Sachin D. Patel, Mark Isalan, Gérald Gavory, Sylvain Ladame, Yen Choo, and Shankar Balasubramanian (2004). „Inhibition of Human Telomerase Activity by an Engineered Zinc Finger Protein that Binds G-Quadruplexe”. Biochemistry. 43 (42): 13452—13458. doi:10.1021/bi048892t.
- ^ Sylvain Ladame, James A. Schouten, Jose Roldan, James E. Redman, Stephen Neidle, and Shankar Balasubramanian (2006). „Exploring the Recognition of Quadruplex DNA by an Engineered Cys2-His2 Zinc Finger Protein”. Biochemistry. 45 (5): 1393—1399. doi:10.1021/bi050229x.
- ^ Bernard A. Brown, Yangqi Li, Julie C. Brown, Charles C. Hardin, John F. Roberts, Stephen C. Pelsue, and Leonard D. Shultz (1998). „Isolation and Characterization of a Monoclonal Anti-Quadruplex DNA Antibody from Autoimmune “Viable Motheaten” Mice”. Biochemistry. 37 (46): 16325—16337. doi:10.1021/bi981354u.
- ^ Christiane Schaffitzel, Imre Berger, Jan Postberg, Jozef Hanes, Hans J. Lipps, and Andreas Plückthun (2001). „In vitro generated antibodies specific for telomeric guanine-quadruplex DNA react with Stylonychia lemnae macronuclei”. PNAS. 98 (15): 8572—8577. doi:10.1073/pnas.141229498.
- ^ Katrin Paeschke, Tomas Simonsson, Jan Postberg, Daniela Rhodes & Hans Joachim Lipps (2005). „Telomere end-binding proteins control the formation of G-quadruplex DNA structures in vivo”. Nature Structural & Molecular Biology. 12: 847 — 854. doi:10.1038/nsmb982.
- ^ Chunying Wei, Guoqing Jia, Jun Zhou, Gaoyi Han and Can Li (2009). „Evidence for the binding mode of porphyrins to G-quadruplex DNA”. Phys. Chem. Chem. Phys. 11: 4025—4032. doi:10.1039/B901027K.
- ^ Richard T. Wheelhouse, Daekyu Sun, Haiyong Han, Frank Xiaoguang Han, and Laurence H. Hurle (1998). „Cationic Porphyrins as Telomerase Inhibitors: the Interaction of Tetra-(N-methyl-4-pyridyl)porphine with Quadruplex DNA” (PDF). J. Am. Chem. Soc. 120: 3261—3262.
- ^ Elzbieta Izbicka, Richard T. Wheelhouse, Eric Raymond, Karen K. Davidson, Richard A. Lawrence, Daekyu Sun, Bradford E. Windle, Laurence H. Hurley, and Daniel D. Von Hoff (1999). „Effects of Cationic Porphyrins as G-Quadruplex Interactive Agents in Human Tumor Cells”. Cancer Res. 59 (2): 639.
- ^ Saurabh Agrawal, Rajendra Prasad Ojha and Souvik Maiti (2008). „Energetics of the Human Tel-22 Quadruplex−Telomestatin Interaction: A Molecular Dynamics Study”. J. Phys. Chem. B. 112 (22): 6828—6836. doi:10.1021/jp7102676.
- ^ Mu-Yong Kim, Hariprasad Vankayalapati, Kazuo Shin-ya, Konstanty Wierzba, and Laurence H. Hurley (2002). „Telomestatin, a Potent Telomerase Inhibitor That Interacts Quite Specifically with the Human Telomeric Intramolecular G-Quadruplex”. J. Am. Chem. Soc. 124 (10): 2098—2099. doi:10.1021/ja017308q.
- ^ Evonne M. Rezler, Jeyaprakashnarayanan Seenisamy, Sridevi Bashyam, Mu-Yong Kim, Elizabeth White, W. David Wilson, and Laurence H. Hurley (2005). „Telomestatin and Diseleno Sapphyrin Bind Selectively to Two Different Forms of the Human Telomeric G-Quadruplex Structure”. J. Am. Chem. Soc. 127 (26): 9439—9447. doi:10.1021/ja0505088.
- ^ Nadrian C. Seeman (1991). „Construction of Three-Dimensional Stick Figures from Branched DNA”. DNA and Cell Biology. 10 (7): 475—486. doi:10.1089/dna.1991.10.475. Проверите вредност параметра
|doi=(помоћ). - ^ Seeman NC (2005). „DNA enables nanoscale control of the structure of matter”. Q. Rev. Biophys. 38 (4): 363—71. PMID 16515737. doi:10.1017/S0033583505004087.
- ^ Collins, M. L.; Irvine, B.; Tyner, D.; Fine, E.; Zayati, C.; Chang, C.; Horn, T.; Ahle, D.; Detmer, J.; Shen, L. P.; Kolberg, J.; Bushnell, S.; Urdea, M. S.; Ho, D. D. (1997). „A branched DNA signal amplification assay for quantification of nucleic acid targets below 100 molecules/ml”. Nucleic Acids Research. 25 (15): 2979—2984. PMC 146852
 . PMID 9224596. doi:10.1093/nar/25.15.2979.
. PMID 9224596. doi:10.1093/nar/25.15.2979.
- ^ Saiki, R.; Scharf, S.; Faloona, F.; Mullis, K.; Horn, G.; Erlich, H.; Arnheim, N. (1985). „Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia”. Science. 230 (4732): 1350—1354. PMID 2999980. doi:10.1126/science.2999980.
- ^ Saiki, R.; Gelfand, D.; Stoffel, S.; Scharf, S.; Higuchi, R.; Horn, G.; Mullis, K.; Erlich, H. (1988). „Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase”. Science. 239 (4839): 487—491. PMID 2448875. doi:10.1126/science.2448875.
- ^ Kevil, C. G., Walsh, L., Laroux, F. S., Kalogeris, T., Grisham, M. B., Alexander, J. S. (1997) An Improved, Rapid Northern Protocol. Biochem. and Biophys. Research Comm. 238:277-279.
- ^ Sandelin, A.; et al. (2007). [ttp://www.nature.com/nrg/journal/v8/n6/abs/nrg2026.html „Mammalian RNA polymerase II core promoters: insights from genome-wide studies”]. Nature Rev. Genet. 8: 424—436.
- ^ Painter PC, Mosher LE, Rhoads C (1982). „Low-frequency modes in the Raman spectra of proteins”. Biopolymers. 21 (7): 1469—72. PMID 7115900. doi:10.1002/bip.360210715.
- ^ Urabe H, Tominaga Y, Kubota K (1983). „Experimental evidence of collective vibrations in DNA double helix (Raman spectroscopy)”. Journal of Chemical Physics. 78 (10): 5937—5939. Bibcode:1983JChPh..78.5937U. doi:10.1063/1.444600.
- ^ Chou KC (1984). „Low-frequency vibrations of DNA molecules”. Biochem. J. 221 (1): 27—31. PMC 1143999
 . PMID 6466317.
. PMID 6466317.
- ^ Chou KC, Maggiora GM, Mao B (1989). „Quasi-continuum models of twist-like and accordion-like low-frequency motions in DNA”. Biophys. J. 56 (2): 295—305. Bibcode:1989BpJ....56..295C. PMC 1280479
 . PMID 2775828. doi:10.1016/S0006-3495(89)82676-1.
. PMID 2775828. doi:10.1016/S0006-3495(89)82676-1.
- ^ Jiang-Cheng Shen, William M. Rideout III and Peter A. Jones (1994). „The rate of hydrolytic deamination of 5-methylcytosine in double-stranded DNA”. Nucl. Acids Res. 22 (6): 972—976. doi:10.1093/nar/22.6.972.
- ^ G. R. Wyatt (1951). „Recognition and estimation of 5-methylcytosine in nucleic acids”. Biochem J. 48 (5): 581—584. Непознати параметар
|pmcid=игнорисан [|pmc=се препоручује] (помоћ) - ^ Wang RY, Kuo KC, Gehrke CW, Huang LH, Ehrlich M (1982). „Heat- and alkali-induced deamination of 5-methylcytosine and cytosine residues in DNA”. Biochimica et Biophysica Acta. 697 (3): 371—7. doi:10.1016/0167-4781(82)90101-4.
- ^ Rudolf Jaenisch & Adrian Bird (2003). „Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals”. Nature Genetics. 33: 245—254. doi:10.1038/ng1089.
- ^ Angela A Andersen, Barbara Panning (2003). „Epigeneticgeneregulation by noncoding RNAs”. Current Opinion in Cell Biology. 15 (3): 281—289. doi:10.1016/S0955-0674(03)00041-3.
- ^ Dana C. Dolinoya, Jennifer R. Weidmana, Randy L. Jirtle (2007). „Epigeneticgeneregulation: Linking early developmental environment to adult disease”. Reproductive Toxicology. 23 (3): 297—307. doi:10.1016/j.reprotox.2006.08.012.
- ^ Wolf Reik (2007). „Stability and flexibility of epigenetic gene regulation in mammalian development”. Nature. 447: 425—432. doi:10.1038/nature05918.
- ^ Bruce K. Duncan, Jeffrey H. Miller (1980). „Mutagenic deamination of cytosine residues in DNA”. Nature. 287: 560 — 561. doi:10.1038/287560a0.
- ^ WM Rideout, 3rd, GA Coetzee, AF Olumi and PA Jones (1990). „5-Methylcytosine as an endogenous mutagen in the human LDL receptor and p53 genes”. Science. 249 (4974): 1288—1290. doi:10.1126/science.1697983.
- ^ Colot V, Rossignol JL (1999). „Eukaryotic DNA methylation as an evolutionary device”. Bioessays. 21 (5): 402—411. PMID 10376011. doi:10.1002/(SICI)1521-1878(199905)21:5<402::AID-BIES7>3.0.CO;2-B.
- ^ Klose R, Bird A (2006). „Genomic DNA methylation: the mark and its mediators”. Trends Biochem Sci. 31 (2): 89—97. PMID 16403636. doi:10.1016/j.tibs.2005.12.008.
- ^ Victoria J. Simpson, Thomas E. Johnson* and Richard F. Hammen (1986). „Caenorhabditis elegans DNA does not contain 5-methylcytosine at any time during development or aging”. Nucl. Acids Res. 14 (16): 6711—6719. doi:10.1093/nar/14.16.6711.
- ^ Bernard Lakowski and Siegfried Hekim (1998). „The genetics of caloric restriction in Caenorhabditis elegans”. PNAS. 95 (22): 13091—13096. doi:10.1073/pnas.95.22.13091.
- ^ Bird A (2002). „DNA methylation patterns and epigenetic memory”. Genes Dev. 16 (1): 6—21. PMID 11782440. doi:10.1101/gad.947102.
- ^ Iqbal, K.; Jin, S. -G.; Pfeifer, G. P.; Szabo, P. E. (2011). „Reprogramming of the paternal genome upon fertilization involves genome-wide oxidation of 5-methylcytosine”. Proceedings of the National Academy of Sciences. 108 (9): 3642—3647. PMC 3048122
 . PMID 21321204. doi:10.1073/pnas.1014033108.
. PMID 21321204. doi:10.1073/pnas.1014033108.
- ^ Dodge JE, Ramsahoye BH, Wo ZG, Okano M, Li E (2002). „De novo methylation of MMLV provirus in embryonic stem cells: CpG versus non-CpG methylation”. Gene. 289 (1–2): 41—48. doi:10.1016/S0378-1119(02)00469-9.
- ^ Haines TR, Rodenhiser DI, Ainsworth PJ (2001). „Allele-Specific Non-CpG Methylation of the Nf1 Gene during Early Mouse Development”. Developmental Biology. 240 (2): 585—598. PMID 11784085. doi:10.1006/dbio.2001.0504.
- ^ Lister R, Pelizzola M, Dowen RH; et al. (2009). „Human DNA methylomes at base resolution show widespread epigenomic differences”. Nature. 462 (7271): 315—22. PMC 2857523
 . PMID 19829295. doi:10.1038/nature08514. Непознати параметар
. PMID 19829295. doi:10.1038/nature08514. Непознати параметар |month=игнорисан (помоћ) - ^ Jaenisch, R.; Bird A. (2003). „Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals”. Nature Genetics. 33 Suppl (3s): 245—254. PMID 12610534. doi:10.1038/ng1089.
- ^ Walsh C, Xu G (2006). „Cytosine methylation and DNA repair”. Curr Top Microbiol Immunol. Current Topics in Microbiology and Immunology. 301: 283—315. ISBN 3-540-29114-8. PMID 16570853. doi:10.1007/3-540-31390-7_11.
- ^ Peter A Jones, Peter W Laird (1999). „Cancer-epigenetics comes of age”. Nature Genetics. 21: 163—167. doi:10.1038/5947.
- ^ Manel Esteller, Paul G. Corn, Stephen B. Baylin, and James G. Herman (2001). „A Gene Hypermethylation Profile of Human Cancer”. Cancer Res. 61: 3225.
- ^ Stephen B Baylin, James G Herman (2000). „DNA hypermethylation in tumorigenesis: epigenetics joins genetics”. 16 (4): 168—174. doi:10.1016/S0168-9525(99)01971-X.
- ^ Craig, JM; Wong, NC (editor) (2011). Epigenetics: A Reference Manual. Caister Academic Press. ISBN 978-1-904455-88-2.
- ^ Smith SS (1998). „Stalling of DNA methyltransferase in chromosome stability and chromosome remodelling”. Int J Mol Med. 1 (1): 147—56.
- ^ Ian M. Smith, Wojciech K. Mydlarz, Suhail K. Mithani, Joseph A. Califano (2007). „DNA global hypomethylation in squamous cell head and neck cancer associated with smoking, alcohol consumption and stage”. International Journal of Cancer. 121 (8): 1724—1728. doi:10.1002/ijc.22889.
- ^ Douglas M. Heithoff, Robert L. Sinsheimer, David A. Low and Michael J. Mahan. „An Essential Role for DNA Adenine Methylation in Bacterial Virulence”. 1999. 284 (5416): 967—970. doi:10.1126/science.284.5416.967.
- ^ Patricia J. Pukkila, Janet Peterson, Gail Herman, Paul Modrich, and Matthew Meselson (1983). „Effects of high levels of DNA adenine methylation on methyl-directed mismatch repair in Escherichia coli”. Genetics. 104 (4): 571—582.
- ^ Kriaucionis S, Heintz N (2009). „The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain”. Science. 324 (5929): 929—30. Bibcode:2009Sci...324..929K. PMC 3263819
 . PMID 19372393. doi:10.1126/science.1169786.
. PMID 19372393. doi:10.1126/science.1169786.
- ^ Ratel D, Ravanat J, Berger F, Wion D (2006). „N6-methyladenine: the other methylated base of DNA”. Bioessays. 28 (3): 309—15. PMC 2754416
 . PMID 16479578. doi:10.1002/bies.20342.
. PMID 16479578. doi:10.1002/bies.20342.
- ^ Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P (1993). „beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei”. Cell. 75 (6): 1129—36. PMID 8261512. doi:10.1016/0092-8674(93)90322-H.
- ^ Padmanava Pradhan, Sampath Tirumala, Xiaohong Liu, Jane M. Sayer, Donald M. Jerina, and Herman J. C. Yeh (2001). „Solution structure of a trans-opened (10S)-dA adduct of (+)-(7S,8R,9S,10R)-7,8-dihydroxy-9,10-epoxy-7,8,9,10-tetrahydrobenzo[a]pyrene in a fully complementary DNA duplex: evidence for a major syn conformation”. Biochemistry. 40 (20): 5870—5881. doi:10.1021/bi002896q.
- ^ Eric J. Schurter, Herman J. C. Yeh, Jane M. Sayer, Mahesh K. Lakshman, Haruhiko Yagi, Donald M. Jerina, David G. Gorenstein (1995). „NMR Solution Structure of a Nonanucleotide Duplex with a dG Mismatch Opposite a 10R Adduct Derived from Trans Addition of a Deoxyadenosine N6-Amino Group to (-)-(7S,8R,9R,10S)-7,8-Dihydroxy-9,10-epoxy-7,8,9,10-tetrahydrobenzo[a]pyrene”. Biochemistry. 34 (4): 1364—1375. doi:10.1021/bi00004a031.
- ^ D. E. Volk, J. S. Rice, B. A. Luxon, H. J. C. Yeh, C. Liang, G. Xie, J. M. Sayer, D. M. Jerina, and D. G. Gorenstein (2000). „NMR Evidence for Syn-Anti Interconversion of a Trans Opened (10R)-dA Adduct of Benzo[a]pyrene (7S,8R)-Diol (9R,10S)-Epoxide in a DNA Duplex”. Biochemistry. 39 (46,): 14040—14053. doi:10.1021/bi001669l.
- ^ B. Epe (1996). „DNA damage profiles induced by oxidizing agents”. Reviews of Physiology Biochemistry and Pharmacology. 127: 223—249. doi:10.1007/BFb0048268.
- ^ Laura J. Kennedy, Kenneth Moore Jr., Jennifer L. Caulfield, Steven R. Tannenbaum, and Peter C. Dedon (1997). „Quantitation of 8-Oxoguanine and Strand Breaks Produced by Four Oxidizing Agents”. Chem. Res. Toxicol. 10 (4): 386—392. doi:10.1021/tx960102w.
- ^ Lawrence J. MarnettCorresponding author contact information (1999). „Lipid peroxidation—DNAdamage by malondialdehyde”. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 424: 83—95. Непознати параметар
|issues=игнорисан (помоћ) - ^ Wei-Xing Zong1, Dara Ditsworth, Daniel E. Bauer, Zhao-Qi Wang, and Craig B. Thompson (2004). „Alkylating DNA damage stimulates a regulated form of necrotic cell death”. Genes & Dev. 18: 1272—1282. doi:10.1101/gad.1199904.
- ^ Raymond R., Gary H.S. Straussb, William P. Peters (1992). „High-dose combination alkylatingagents with autologous bone-marrow support in patients with breast cancer: preliminary assessment of DNAdamage in individual peripheral blood lymphocytes using the single cell gel electrophoresis assay”. Mutation Research/Environmental Mutagenesis and Related Subjects. 271 (2): 101—113.
- ^ Silvia Tornaletti, Gerd P. Pfeifer (1995). „UV Light as a Footprinting Agent: Modulation of UV-induced DNADamage by Transcription Factors Bound at the Promoters of Three Human Genes”. Journal of Molecular Biology. 249 (4): 714—728.
- ^ C Kielbassa, L Roza and B Epe (1997). „Wavelength dependence of oxidative DNA damage induced by UV and visible light”. Carcinogenesis. 18 (4): 811—816. doi:10.1093/carcin/18.4.811.
- ^ Kai Rothkamm and Markus Löbrich (2003). „Evidence for a lack of DNA double-strand break repair in human cells exposed to very low x-ray doses”. PNAS. 100 (9): 5057—5062. doi:10.1073/pnas.0830918100.
- ^ Timothy J. Kinsella, Patricia P. Dobson, James B. Mitchell, Albert J. Fornace Jr. (1987). „Enhancement of Xray induced DNAdamage by pre-treatment with halogenated pyrimidine analogs”. International Journal of Radiation. 13 (5): 733—739.
- ^ Douki T, Reynaud-Angelin A, Cadet J, Sage E (2003). „Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation”. Biochemistry. 42 (30): 9221—6. PMID 12885257. doi:10.1021/bi034593c.
- ^ Lawrence J. Marnett (2000). „Oxyradicals and DNA damage”. Carcinogenesis. 21 (3): 361—370. doi:10.1093/carcin/21.3.361.
- ^ Susan S. Wallace (1998). „Enzymatic Processing of Radiation-Induced Free Radical Damage in DNA”. Radiation Research. 150 (5s): S60—S79.
- ^ Matthews O Bradleya, Leonard C Erickson (1981). „Comparison of the effects of hydrogen peroxide and X-ray irradiation on toxicity, mutation, and DNAdamage/repair in mammalian cells (V-79)”. Biochimica et Biophysica Acta (BBA) - Nucleic Acids and Protein Synthesis. 654 (1): 135—141.
- ^ JA Imlay, SM Chin and S Linn (1988). „Toxic DNA damage by hydrogen peroxide through the Fenton reaction in vivo and in vitro”. Science. 240 (4852): 640—642. doi:10.1126/science.2834821.
- ^ Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S (1999). „Hydroxyl radicals and DNA base damage”. Mutat Res. 424 (1–2): 9—21. PMID 10064846. doi:10.1016/S0027-5107(99)00004-4.
- ^ Beckman KB, Ames BN (1997). „Oxidative decay of DNA”. J. Biol. Chem. 272 (32): 19633—6. PMID 9289489. doi:10.1074/jbc.272.32.19633.
- ^ Valerie K, Povirk L (2003). „Regulation and mechanisms of mammalian double-strand break repair”. Oncogene. 22 (37): 5792—812. PMID 12947387. doi:10.1038/sj.onc.1206679.
- ^ Ferguson L, Denny W (1991). „The genetic toxicology of acridines”. Mutat Res. 258 (2): 123—60. PMID 1881402.
- ^ Stephens T, Bunde C, Fillmore B (2000). „Mechanism of action in thalidomide teratogenesis”. Biochem Pharmacol. 59 (12): 1489—99. PMID 10799645. doi:10.1016/S0006-2952(99)00388-3.
- ^ Toufan Parman, Michael J. Wiley & Peter G. Wells (1999). „Free radical-mediated oxidative DNA damage in the mechanism of thalidomide teratogenicity”. Nature Medicine. 5: 582 — 585. doi:10.1038/8466.
- ^ Jeffrey A (1985). „DNA modification by chemical carcinogens”. Pharmacol Ther. 28 (2): 237—72. PMID 3936066. doi:10.1016/0163-7258(85)90013-0.
- ^ Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A (2001). „Intercalators as anticancer drugs”. Curr Pharm Des. 7 (17): 1745—80. PMID 11562309. doi:10.2174/1381612013397113.
Литература
- Dumanović, J, marinković, D, Denić, M: Genetički rečnik, Beograd, 1985.
- Kosanović, M, Diklić, V: Odabrana poglavlja iz humane genetike, Beograd, 1986.
- Lazarević, M: Ogledi iz medicinske genetike, Beograd, 1986.
- Marinković, D, Tucić, N, Kekić, V: Genetika, Naučna knjiga, Beograd
- Matić, Gordana: Osnovi molekularne biologije, Zavet, Beograd, 1997.
- Ridli, M: Genom - autobiografija vrste u 23 poglavlja, Plato, Beograd, 2001.
- Tatić, S, Kostić, G, Tatić, B: Humani genom, ZUNS, Beograd, 2002.
- Tucić, N, Matić, Gordana: O genima i ljudima, Centar za primenjenu psihologiju, Beograd, 2002.
- Šerban, Nada: ćelija - strukture i oblici, ZUNS, Beograd, 2001
- Berry, Andrew; Watson, James D (2003). DNA: the secret of life. New York: Alfred A. Knopf. ISBN 0-375-41546-7.
- Calladine, Chris R.; Drew, Horace R.; Luisi, Ben F. and Travers, Andrew A. (2003). Understanding DNA: the molecule & how it works. Amsterdam: Elsevier Academic Press. ISBN 0-12-155089-3.
- Dennis, Carina; Julie Clayton (2003). 50 years of DNA. Basingstoke: Palgrave Macmillan. ISBN 1-4039-1479-6.
- Horace Freeland Judson 1979. The Eighth Day of Creation: Makers of the Revolution in Biology. Touchstone Books, ISBN 0-671-22540-5. 2nd edition: Cold Spring Harbor Laboratory Press, 1996 paperback: ISBN 0-87969-478-5.
- Olby, Robert C. (1994). The path to the double helix: the discovery of DNA. New York: Dover Publications. ISBN 0-486-68117-3., first published in October 1974 by MacMillan, with foreword by Francis Crick;the definitive DNA textbook,revised in 1994 with a 9 page postscript
- Micklas, David. 2003. DNA Science: A First Course. Cold Spring Harbor Press: ISBN 978-0879696368
- Ridley, Matt (2006). Francis Crick: discoverer of the genetic code. Ashland, OH: Eminent Lives, Atlas Books. ISBN 0-06-082333-X.
- Olby, Robert C. (2009). Francis Crick: A Biography. Plainview, N.Y: Cold Spring Harbor Laboratory Press. ISBN 0-87969-798-9.
- Rosenfeld, Israel. 2010. DNA: A Graphic Guide to the Molecule that Shook the World. Columbia University Press: ISBN 978-0231142717
- Schultz, Mark and Zander Cannon. 2009. The Stuff of Life: A Graphic Guide to Genetics and DNA. Hill and Wang: ISBN 0809089475
- Gunther Stent Siegmund; James D. Watson (1980). The double helix: a personal account of the discovery of the structure of DNA. New York: Norton. ISBN 0-393-95075-1.
- James D. Watson 2004. DNA: The Secret of Life. Random House: ISBN 978-0099451846
- Maurice Wilkins (2003). The third man of the double helix the autobiography of Maurice Wilkins. Cambridge, Eng: University Press. ISBN 0-19-860665-6.
Спољашње везе
- Бионет школа
- ДНК на сајту Curlie
- Предвиђање ДНК места везивања на протеину
- ДНК намотавање да би се формирали хромозоми
- Игра ДНК двоструког хеликса
Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA
Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA